서론
마디풀과(Polygonaceae)는 전 세계에 약 40속 1,100여종이 분포하며(Brandbyge, 1993; Reveal, 1989; Thorne, 2000), 우리나라에는 7속 75종이 생육하고 있는 것으로 알려져 있다(Park and Hong, 2007). 우리나라 농경지에 발생하는 잡초는 81과 619종으로 이 중 마디풀과는 29분류군이 분포하여 전체 잡초 중 상위에서 다섯 번째로 점유율이 높은 잡초이다. 주로 개여뀌(Persicaria longiseta Bruijn), 소리쟁이(Rumex crispus L.), 여뀌(P. hydropiper (L.) Delarbre), 봄여뀌(Persicaria vulgaris Webb & Moq.), 마디풀(Polygonum aviculare L.), 고마리(Persicaria thunbergii Siebold & Zucc. H. Gross), 돌소리쟁이(R. obtusifolius L.) 등 7-8종이 주를 이루고 있다(Lee et al., 2017; Park et al., 2001; Park et al., 2003).
잡초관리는 고품질의 농산물 생산과 농촌 노동력 절감을 위하여 필수적이다. 정확하게 잡초종을 동정하면 종에 따라 적절한 경종적, 물리적, 화학적 방제 방법을 선정하는 등 잡초를 효율적으로 관리하는데 도움이 된다. 일부 종들은 전문가의 도움 없이 정확하게 동정하기 매우 어려움에도 불구하고, 전 세계적으로 형태분류학자는 감소추세에 있기 때문에 종을 정확하고 빠르게 동정하는 것은 점점 더 어려워지고 있다. 또한 분자생물학적 기법을 이용한 분류 결과, 과거에 형태적으로 다른 종으로 분류되었던 종들이 이명으로 처리되거나 또는 종 복합체인 것으로 밝혀지고 있어 단순한 동정은 더욱 어려운 실정이다
Hebert et al. (2003)에 의해 제안된 DNA 바코드는 게놈의 짧은 표준 범위를 이용하여 형태적 분류에 대한 지식이 없어도 종을 빠르고 쉽고 정확하게 동정할 수 있는 방법이다. 최근에 형태분류학자의 부재 하에 형태학적으로 동정하기 어려운 분류군을 동정하는 대안으로 활발하게 이용되고 있으며 각 국은 자국의 종들에 대하여 바코드 데이터베이스를 구축하여 생물다양성 보전에도 주의를 기울이고 있다. 동물에서는 미토콘드리아 CO1 (Cytochrome Oxidase 1) 유전자를 DNA 바코드 표준 유전자로 이용하고 있으나 고등식물에서는 552종의 72% 이상을 동정해낼 수 있는 엽록체 DNA의 rbcL과 matK 조합의 이용을 권고하고 있다(CBOL Plant Working Group, 2009). 또한 분류군에 따라 핵산 DNA의 ITS(internal transcribed spacer)나 엽록체 DNA의 psb A-trn H 등 추가 마커를 활용하고 있다(Chen et al., 2010; Lee et al., 2015a; Lee et al., 2015b).
본 연구는 국내 농경지에서 흔하게 발생하는 마디풀과 잡초를 대상으로 엽록체 DNA의 rbcL과 matK 부위를 바코드하고 그 결과를 미국 국립생물공학정보센터(National Center for Biotechnology Information, NCBI)에 등록하여 일반인들이 마디풀과 잡초 동정에 DNA 바코드 정보를 쉽게 이용할 수 있는 기초 자료를 만들고자 하였다.
재료 및 방법
발아생리
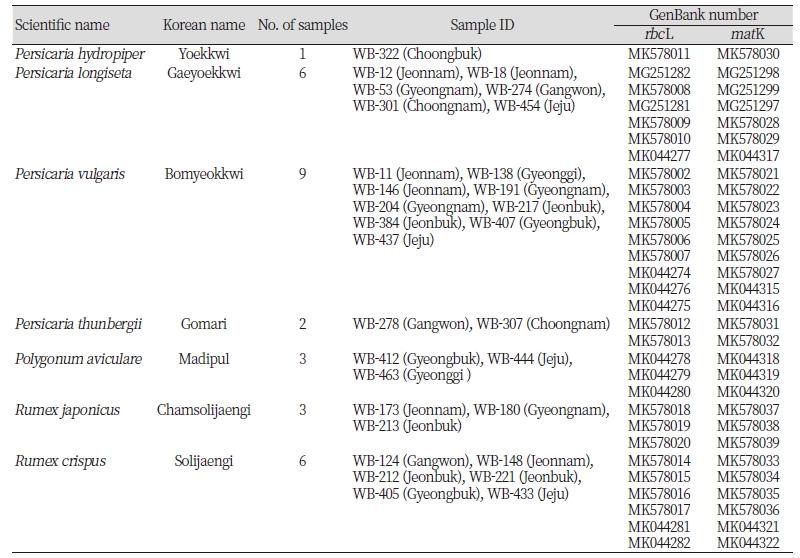
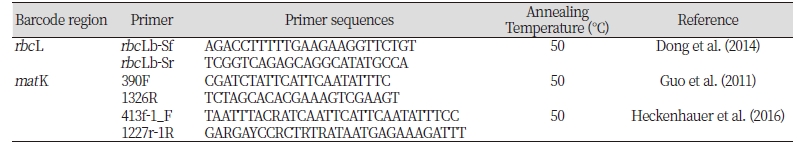
경기도, 강원도, 충청도, 전라도, 경상도, 제주도 등 전국의 농경지에서 마디풀과 7분류군 30생태형을 채집하여 엽록체 DNA rbcL과 matK 부위를 이용하여 바코드 하였다(Table 1). 바코드 분석을 위하여 DNA는 DNeasy Plant Mini Kit (Qiagen, USA)를 이용하여 제공되는 방법에 따라 분리하였다. PCR 증폭반응은 genomic DNA 0.5 μL, 10 mM의 dNTP 0.4 μL, 25 mM의 MgCl 1 μL, 5x GotaqⓇ Flexi Buffer 4 μL, 4 pmole의 rbcL/matK forward, reverse primer 1 μL씩 그리고 0.5 unit의 taq DNA polymerase 0.1 μL를 혼합하여 수행하였다. 증폭반응은 T-100 thermal cycle (BIO-RAD)를 이용하여 95℃에서 5분간 pre-denaturation 후, 95℃에서 1분간 denaturation, 증폭 조건에 따라 48 또는 50℃에서 1분간 annealing 그리고 72℃에서 1분간 extension을 35회 수행하였다(Table 2). 증폭 산물은 QIAGEN QIAquickⓇ PCR Purification Kit (Qiagen, USA)를 이용하여 제공되는 방법에 따라 정제하였다.
PCR과 염기서열 분석에 이용된 마커는 Table 2와 같으며, 염기서열의 분석은 GenoTech (Dajeon, Korea)에서 수행하였다. 분석된 염기서열은 Lasergen 8.1.5 (CodonCode Corp, Dedham, USA)를 이용하여 contig를 작성하고 BioEdit 7.2.6의 Clustal W를 이용하여 정렬한 후 육안 편집을 거쳤다. 본 연구에서 분석된 염기서열을 비교하기 위하여 미국 국립생물공학정보센터(National Center for Biotechnology Information, NCBI)에서 마디풀과의 rbcL과 matK 염기서열을 내려 받았으며, 내려 받은 염기서열은 MEGA 6.06 (Tamura et al., 2013) 프로그램에서 Clustal W를 이용하여 본 시험에서 분석된 염기서열과 함께 정렬하였다. Kimura 2-Parameter (K2P) distance model 옵션으로 종내ㆍ종간 변이를 산출하고, Neighborjoining 계통도를 작성하였다(Saitou and Nei, 1987).
본 바코드에 이용된 마디풀과 잡초의 확증표본을 제작하였으며 표본은 국립농업과학원 표본관(HCCN)에 보관하였다. 분석된 rbcL과 matK 바코드 염기서열은 NCBI에 제출하여 GenBank 번호를 부여 받았다.
결과 및 고찰
각 마커별 바코드 염기서열은 미국 국립생물공학정보센터에 제출하여 GenBank 번호를 받아 공개하였으며 GenBank번호는 Table 1과 같다.
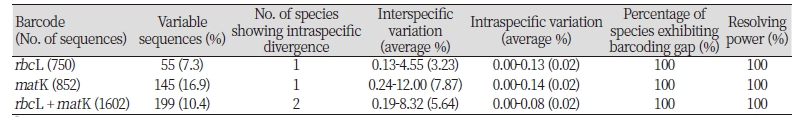
마디풀과의 각 마커별 염기서열 분석 성공률은 rbcL과 matK 모두 100% 이었다. rbcL은 단일 프라이머와 단 한 번의 시도로 PCR 증폭과 염기서열 분석이 성공적으로 진행된 반면, matK는 분류군에 따라서 2회 이상의 PCR을 시도하여 성공 하였다. 그러나 벼과의 rbcL 88.7%와 matK 73.7%의 성공률(Lee et al., 2015b)에 비하여 현저히 높은 성공률을 보였으므로 이 두 마커는 마디풀과의 동정에 추가 마커 없이 사용될 수 있을 것으로 판단된다. matK는 종간에 높은 변이율을 보유하 여 분자계통학적 연구에 유용하게 이용되고 있지만 Kress and Erickson (2007)은 matK의 PCR 증폭률이 낮고(39.3%), 변이에 의해 보존된 염기서열이 부족하기 때문에 프라이머를 제작하는데 어려움이 있어 matK를 육상식물의 바코드 마커 로 이용하기에 부족하다고 보고한 바 있다. 그러나, 마디풀과에서는 무난하게 적용 가능하였으며, 분류군별로 필요 시 적절한 추가 마커를 사용하면 정확한 바코드에 더 도움이 될 것으로 생각한다.
각 바코드 마커의 염기서열 변이는 rbcL에서 7.3%, matK에서 16.9%로 matK의 염기서열 변이가 높았다. rbcL의 고마리와 matK의 개여뀌는 종내 변이를 보였으며 이들의 종내 변이율은 모두 평균 0.02%로 차이가 없었다. 두 마커의 조합 인 rbcL + matK에서도 0.02%의 종내 변이율을 보였다(Table 3). 봄여뀌와 소리쟁이, 참소리쟁이(Rumex japonicus Houtt.)는 rbcL과 matK에서 종내 변이를 보이지 않았다. 종내 변이를 나타내지 않는 이유는 국내에서는 한 가지 생태형만이 존재하거나, 샘플링의 수가 적어서 다양한 스펙트럼의 변이를 확인하지 못했기 때문일 것이다. 그러므로 본 연구에서 종 내 변이를 나타내지 않은 종들은 국내에 분포하는 단일 생태형 여부를 확인하기 위하여 추후 더 많은 지역의 샘플을 포 함하여 분석할 필요가 있다.
단일 마커의 종간 변이율은 rbcL에서 3.23%, matK에서 7.87% 이었으며, rbcL + matK조합에서 5.64%를 보였다(Table 3). rbcL 바코드마커를 이용한 분석에서는 마디풀이 봄여뀌와 가장 종간 변이가 큰 반면, matK에서는 마디풀과 여뀌의 종간변이가 가장 큰 것으로 파악되었다.
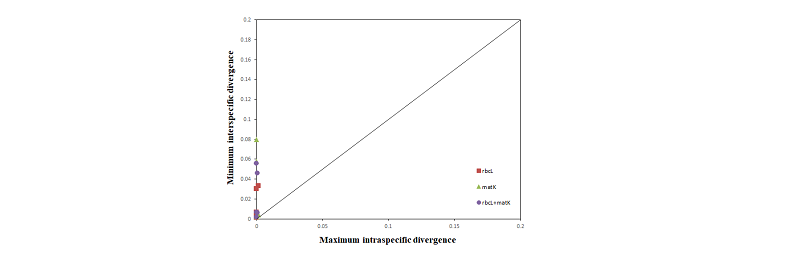
두 개체 이상의 생태형을 갖는 분류군의 최대 종내 변이와 최소 종간 변이의 차이가 0보다 클 때 바코드 갭이 있는 것으로 정의하며, 다른 종과는 바코드 갭이 있어야 한다(Theodoridis et al., 2012). 본 연구에 이용된 마디풀과는 rbcL과 matK, rbcL + matK 모두 100%의 바코드 갭을 갖는 것으로 분석되어(Table 3; Fig. 1) 바코드 마커를 이용한 종 동정은 정확한 것으로 생각한다.
Neighbor-joining 계통수의 단일계통 형성여부에 근거한 바코드 해상력 또한 바코드 캡과 마찬가지로 rbcL과 matK, rbcL + matK 모두 100%로 분석되어, rbcL과 matK의 단일 neighbor-joining 계통수와 이들 조합의 계통수 모두 마디풀과의 분류군들을 구분하는데 성공하였다(Table 3). Kim et al. (2008)에 의하면 여뀌속은 잎의 형태에 따라 이형인 고마리, 타원형 또는 피침형을 동시에 또는 각각 보이는 여뀌, 봄여뀌, 개여뀌로 구분 할 수 있다. 여뀌, 봄여뀌, 개여뀌의 경우는 비슷한 잎의 형태를 가지고 있어 옆신의 표면 중앙부의 흑색 반문이 유무에 따라 있는 경우 봄여뀌, 개여뀌로 없는 경우 여뀌로 구분하며, 탁엽 끝의 털의 길이가 탁엽보다 짧으면 봄여뀌로 탁엽과 비슷하거나 더 길면 개여뀌로 구분 할 수 있다.
Song (2001)은 소리쟁이속은 수과를 둘러싸는 내화피편이 전연일 경우 소리쟁이로 예거치일 경우 참소리쟁이로 구분할 수 있다고 하였다. 그러나 여뀌속이나 소리쟁이속 모두 형태적으로 형질의 차이가 쉽게 구분되지 않거나 연속변이가 관찰되어 정확하게 동정하기에 어려움이 있다. 여뀌속이나 소리쟁이속에서 동정이 의심되는 생태형이 발견될 경우, 바코드를 활용하면 더 정확한 종 동정을 할 수 있을 것으로 생각한다. 실제로 현장에서 돌소리쟁이(WB-213)로 동정한 분류군이 본 바코드 결과 참소리쟁이(Rumex japonicus Houtt.)로 동정이 되었으며, 봄여뀌에서 개여뀌(WB-191)로 동정되거나 개여뀌에서 봄여뀌(WB-407)로 동정이 되는 경우도 있었다.
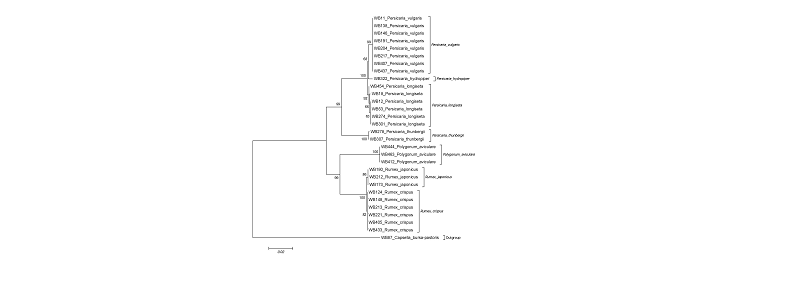
Fig. 2. Neighbor-joining tree inferred from the analysis of rbcL + matK from Polygonaceae. Branch lengths are proportional to distances estimated from the two-parameter method of KIMURA. Numbers above the branch indicates the bootstrap supports (%) for 1000 replicated analyses. Capsella bursa-pastoris was used as outgroup.