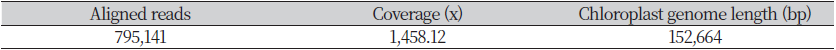서 언
가시상추(Lactuca serriolaL)는 유럽 원산의 국화과 일년생 식물이다. 국내에서는 일반적으로 10~12월에 발아하고 로제트형태로 월동하여 이듬해 7월경에 꽃이 피는 동계일년생 잡초의 생활환(life cycle)을 가지고 있지만, 4~5월에 발아하여 7월경에 꽃이 피는 하계일년생잡초의 생활사를 보이기도 한다(Prince et al., 1978). 우리나라에서는 1980년에 김포공항 및 구마고속도로에 분포한다고 최초로 보고된 것으로 봤을 때(Yim and Jeon, 1980), 가시상추의 국내 유입은 1980년도 이전일 것으로 판단하고 있다. 높은 환경적응력과 빠른 확산성 등을 이유로 가시상추는 2012년에 환경부에서 생태계교란생물(Invasive Alien Species)로 지정되었다(Kim et al., 2017). 국내에서는 주로 도로변, 하천변, 나지 등 인간의 개입이 비교적 적은 곳에 분포하고 있으며, 비교적 빠른 시간에 군락을 이룬다(Kim et al., 2013).
가시상추는 방제가 쉽지 않은 잡초로 알려져 있으며, 이미 제초제 저항성도 보고되어 있다. 미국 아이다호에서 설포닐유레아(sulfonylurea) 및 이미다졸리논(imidazolinone)계열의 제초제 저항성 가시상추가 최초로 보고된 이 후(Mallory-Smith et al., 1990), 호주에서도 acetolactate synthase 저해 제초제에 저항성이 보고되고 있으며(Preston et al., 2006), 미국 워싱턴에서는 합성옥신계 제초제(2,4-D, dicamba 및 MCPA) 저항성 가시상추가 보고되기도 하였다(Burke et al., 2009). 최근 국내에서도 가시상추가 과수원 등 농경지로 확산되고 있으며(Kim et al., 2017), 이를 방제하기 위하여 다양한 연구가 진행되고 있다. 특히 가시상추 기주 특이적 병원균을 활용한 생물학적 방제법 개발이 기대되고 있다(Kim and Choi, 2020).
초기 유입단계의 생태계교란식물은 선제적으로 강력하게 방제를 해야한다(Ruesink et al., 1995). 그러나 이미 확산단계에 이른 생태계교란식물은, 남아프리카공화국에서 Acacia spp.의 방제 사례처럼, 원산지 및 유전적/생태적 특성을 파악한 후 천적 등 생물학적 방제원(biological control agent)을 찾는 것이 더 효율적이라고 판단하고 있다(Zachariades et al., 2017). 즉, 생태계교란식물의 원산지 정보는 생물학적 방제원을 선발함에 있어 매우 중요하며, 전제되어야 할 정보로 여기고 있다. 생태계교란종의 원산지 추적은 주로 예상 원산지 혹은 예상 유입경로 지역 수집종과 현지 수집종의 분자생물학적 정보를 비교하며 이루어진다. 특정 유전자 내 염기서열을 비교할 수도 있으며(Lees et al., 2011), RAPD (randomly amplified polymorphic DNA), SSR (simple sequence repeat) 등의 방법을 활용할 수도 있다(Hirsch et al., 2011, Roché et al., 2003).
본 연구는 SSR 마커를 이용해 국내 및 해외의 가시상추 집단의 유전적 다양성을 확인하여 국내 분포 가시상추의 원산지 혹은 유입경로를 추적하고자 하였으며, 이를 통해 가시상추 생물학적 방제의 기초자료로 활용하고자 하였다.
재료 및 방법
식물재료 및 DNA 추출
본 연구에서 사용한 가시상추는 총 14지역에서 수집하였다(Table 1). 국내 가시상추는 총 7지역(경북 안동, 충북 청주, 전북 김제, 전남 광양, 대구, 부산, 및 충남 당진)에서 군락을 확인하였으며, 약 1 m 간격으로 각 군락 당 10개체를 채집하였다. 해외 가시상추 수집종은 미국 종자은행(Plant Germplasm Inspection Station, USDA-ARS) 에서 체코, 네덜란드, 중국, 불가리아, 조지아, 아프가니스탄 및 포르투갈 이상 7개국의 가시상추 종자를 확보하였으며(Table 1), 국내에서 생육을 통해 각각 8개체를 채집하였다. 채집한 수집종은 액체질소와 함께 막자사발에서 곱게 갈았으며, 모든 가시상추 수집종의 DNA는 Dneasy® plant mini kit (QIAGEN, Hilden, Germany)를 이용하여 제공되는 방법에 따라 추출하였다.
de novo assembly 및 염기서열 분석
가시상추의 엽록체 유전체 (chloroplast genome; cpDNA)의 염기서열을 분석하기 위해 충북 충주에서 수집한 가시상추(chungju4)의 잎에서 DNA를 앞서 기술한 바와 같은 방법으로 추출하였다. NGS sequencing은 Illumina MiSeq platform (Illumina Inc., CA, USA)을 사용하였으며, CLC Assembly Cell package ver. 4.2.1 (QIAGEN, Hilden, Germany)를 이용하여 de novo assembly를 수행하였다. Contig 중 엽록체 유전체 유래 contig만을 선별하여 de novo assembly of low coverage whole genome shotgun sequencing (dnaLCW) 방법으로 cpDNA 유전자 지도를 완성하였다. dnaLCW 방법은 whole genome sequencing으로 생산된 대량의 염기서열 정보 중 핵 유전체 크기의 0.5 ~ 1X에 해당하는 low coverage NGS data를 이용해 세포소기관의 유전체 서열을 완성하고 정보를 분석하는 방법이다(Kim et al., 2015). Mega BLAST search을 통해 가시상추와 homology가 가장 높은 종의 cpDNA 염기서열을 확인하고 BLASTZ 를 이용하여 유전체 구조를 비교하였다(Schwartz et al., 2003).
SSR 마커 선발
EST-SSR 마커: 기 발표된 문헌을 바탕으로 가시상추의 다형성을 검정할 수 있는 104개의 EST-SSR 마커를 확인하였다(data not shown). 국내 및 해외 종의 DNA에 대한 각 마커의 PCR 및 다형성 검정 등을 바탕으로 최종 10개의 EST-SSR 마커를 가시상추의 다형성 검정에 사용하였다(Table 2).
|
Table 2. Information of primers used in this study, EST-SSR markers and developed markers based on chloroplast genome sequence analysis. 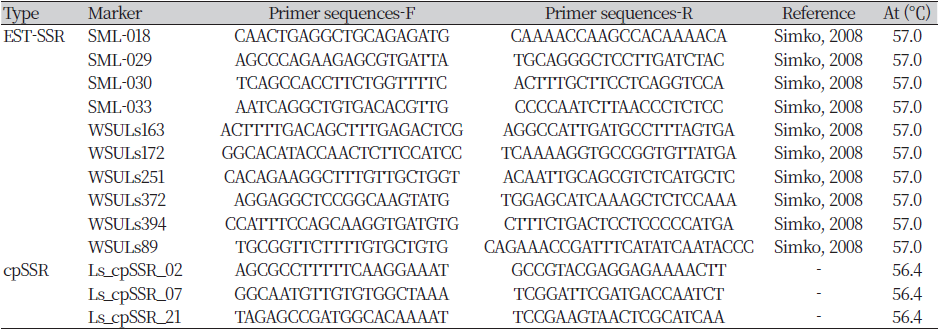
|
cpDNA-SSR 마커: MISA-Web (https://webblast.ipk-gatersleben.de/misa/index.php)을 이용해 가시상추 엽록체 유전체 내 SSR의 분포를 탐색하였다. 총 123개의 엽록체 내 SSR 중, SSR의 종류 및 motif의 길이 등을 바탕으로 36개를 1차 선발한 뒤, PCR 및 다형성 검정 등을 통해 최종적으로 총 3개의 마커를 선발하였다(Table 2).
다형성 검정
13개의 SSR마커를 annealing temperature (At) 및 primer dimer 생성 가능성을 고려하여 2개씩 짝지어 총 7개의 마커그룹을 만들었다. 그룹 내 두 개의 forward primer에 각각 6-carboxyfluorescein (FAM) 및 hexachloro-fluoresceine (HEX)의 형광표지(Fluorescence tag)를 붙였다. 앞서 추출한 가시상추 gDNA (10 ng μL-1) 1 μL, FAM 및 HEX로 형광표지된 forward primer (5 ρM) 0.6 μL, reverse primer (5 ρM) 0.6 μL, 10X buffer 1.25 μL, dNTPs (2 mM each) 1.25 μL, 및 1.25 units F-Star Taq polymerase (Biofact, Daejeon, Korea)를 혼합하여 총 12.5 μL의 PCR cocktail을 제조하였다. PCR 조건은 다음과 같다. 95℃에서 10분동안 initial denaturation하고, 총 2분(95℃에서 30초, 58℃에서 30초, 72°C에서 1분)을 30회 반복한 후, 68℃에서 30분동안 final extension하였다. PCR 산물은 ABI3730xl DNA analyzer (Applied Biosystems, Foster, USA)를 사용하여 전기영동한 후, GeneMapper 4.0 (Applied Biosystems, Foster, USA)을 이용하여 대립유전자의 크기를 분석하였다. 주요대립유전자의 출현빈도(major allele frequency), 대립유전자 수(number of alleles; NA), gene diversity (GD), 이형접합성(heterozygosity), and polymorphism information content (PIC) 등은 PowerMarker v.3.25를 사용하여 분석하였다(Liu and Muse, 2005). F-statistics 및 주성분분석(principal coordinates analysis; PCoA)은 GenAlEx 6.501을 통해 분석하였다(Peakall and Smouse, 2006).
결과 및 고찰
de novo assembly 및 염기서열 분석
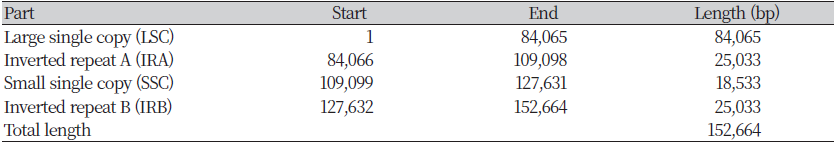
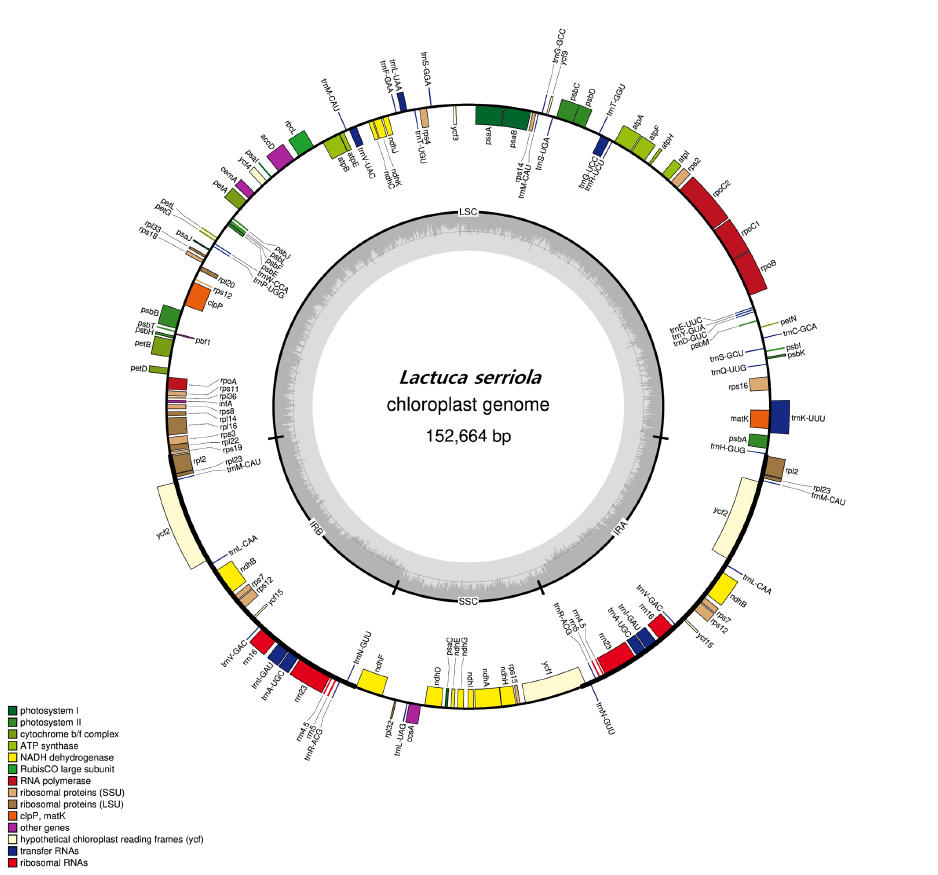
완성된 가시상추 cpDNA 염기서열의 길이는 152,664 bp인 것으로 조사되었다(Table 3). Aligned read는 795,141개였으며, average coverage는 1458.12X였다(Table 3). dnaLCW 방법으로 가시상추 cpDNA를 assembly한 결과, large single copy (LSC, 84,065 bp), inverted repeat A (IRA, 25,033 bp), small single copy (SSC, 18,533 bp), inverted repeat (IRB, 25,033 bp)가 이어지는 circular 형태의 구조로 분석되었다(Table 4; Fig. 1). 완성된 가시상추의 엽록체 염기서열의 mega BLAST 결과, 가장 상동성(homology)이 높은 종은 상추(Lactuca sativa)였으며, 국화과 잡초인 이고들빼기(Youngia denticulata) 및 뽀리뱅이(Y. japonica)가 그 뒤를 이었다.
가시상추의 cpDNA는 총 112개의 유전자로 구성되어 있는 것으로 분석되었다(Suppl. Table 1). Protein-coding genes (PCGs)는 80개였고, transfer RNA (tRNA)는 28개였으며, ribosomal RNA (rRNA)는 4개였다. 이 중, 18개의 유전자(rps7, rps12, ndhB, rpl2, rpl23, rrn4.5, rrn5, rrn16, rrn23, ycf2, ycf15, trnA-UGC, trnI-GAU, trnL-CAA, trnM-CAU, trnN-GUU, trnR-ACG, trnV-GAC)가 2 copy 이상 존재하고 있었다. IRA 및 IRB는 PCGs 7개, tRNA 7개, 그리고 rRNA 4개가 반복되어 있는 것으로 분석되었다.
다형성 검정
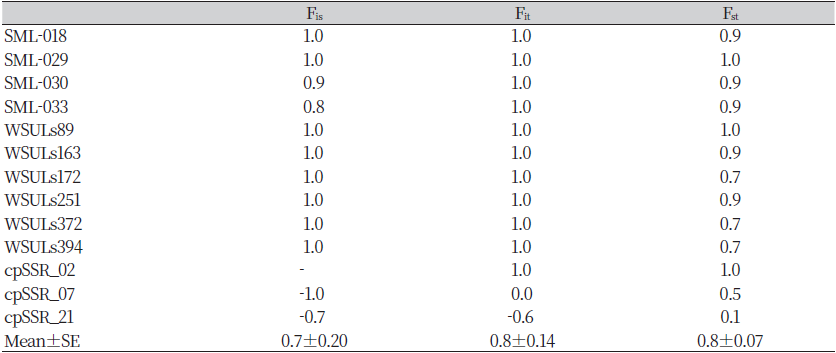
전체 14개 population에 대한 13개 마커의 다형성 검정결과, 대립유전자의 수(No. of different alleles)는 1.3±0.05 였으며, 유효대립유전자 수(No. of effective alleles)는 1.2±0.04이었다(Table 5). 관측 이형접합성(Observed heterozygosity, HO)과 기대 이형접합성(Expected heterozygosity, HE)은 각각 0.1±0.02 및 0.1±0.02였다. 전체적으로 HE와 HO가 낮고 차이가 거의 없어(Table 5), 집단 내 변이가 거의 없는 것으로 판단되었다. F-statistics 분석 결과, 전체적으로 Fst값, 이 0.8±0.07로 매우 높았으며, Fis 및 Fit값또한 1에 가까웠다(Table 6). 이와 같은 결과들을 종합해 볼 때, 실험에 사용한 집단들은 유전적으로 고정되어 있다고 판단할 수 있으며, 집단 내에서 유전적 다양성이 매우 낮은 특징을 보인다고 할 수 있다. 원산지로 알려진 유럽 내 가시상추 집단의 유전적 다양성을 비교한 연구에서도 비교적 낮은 유전적 다양성을 보였다(van de Wiel et al., 2010). 이러한 낮은 유전적 다양성은 두 가지로 해석할 수 있다.
|
Table 5. No. of alleles, observed and expected heterozygosity, and fixation index of 13 markers used in this study. 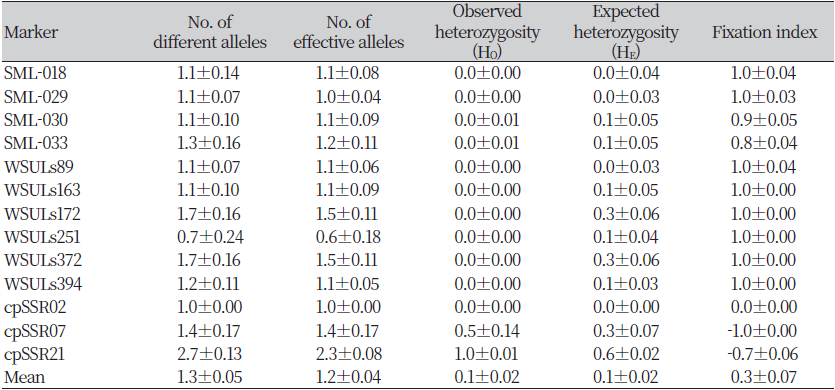
|
Vavilov의 유전자중심설에 따르면, 식물의 원산지에 가까울수록 대상 식물의 유전적 다양성이 크며, 원산지에서 멀어질수록 변이가 적다(Vavilov et al., 1992). 또한, 원산지에서 멀어질수록 생육에 불리한 환경에 적응하기 위해 유전적 다양성은 낮아질 수도 있다(Lebeda et al., 2009). 우리나라는 가시상추의 원산지로 알려진 지중해 연안의 반대편에 있으며 겨울은 더 혹독하다. 게다가 국내에서 가시상추 집단이 형성된 곳은 주로 극심한 수분스트레스가 있는 나지나 도로변, 혹은 제방이다(Kim et al., 2013). 우리나라의 환경은 원산지와 비교했을 때 유전적으로 다양한 가시상추가 적응할 수 있는 기후나 환경은 아니라는 점으로 국내 가시상추 수집종의 유전적 다양성이 낮은 이유를 설명할 수 있을 것이다.
가시상추의 수분특성(pollination characteristic)도 국내 가시상추 수집종의 유전적 다양성이 낮은 이유를 설명할 수 있다. 가시상추는 자가수분 식물이며, 다른 종과의 종간교잡의 비율 또한 낮은 것으로 알려져 있다(Lu et al., 2007, Mejías, 1994). 대표적인 선구식물이며, 많은 종자 생산량 및 낮은 휴면성으로 인해 빠르게 확산하는 특징을 보인다. 우리나라 유입 시기가 사회적으로 인적/물적 교류가 활발해지기 시작한 1980년대 직전이라는 점과 발생 초기에는 주로 경상도 지역을 중심으로 분포했다가 경기도 등 전국으로 확산했다는 점을 봤을 때(Kim et al., 2013), 다양한 종류의 가시상추가 다양한 경로를 통해 대량으로 유입되었을 가능성보다 국내 환경에 적응한 일부 개체를 중심으로 확산했다고 보는 것이 타당할 것으로 보인다. 자가수분식물인 가시상추의 확산 경향으로 판단했을 때, 국내 가시상추 수집종 간의 유전적 다양성이 낮은 것은 그 자체의 생물학적인 요인도 포함한다고 할 수 있을 것이다.
주성분분석을 통한 국내 가시상추 유입경로 추적
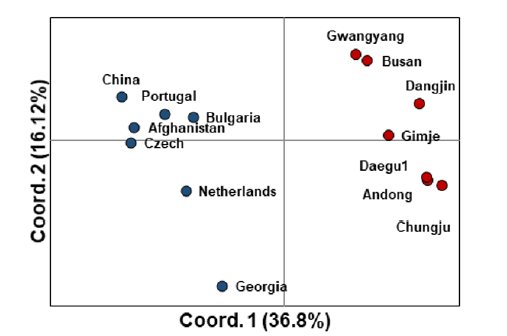
국내 가시상추 7 집단 및 해외 가시상추 7집단에 대한 SSR 마커 13종의 다형성을 기반으로 주성분분석한 결과는 Fig. 2와 같았다. 제1주성분으로 36.8%를 설명할 수 있었으며, 제2주성분으로 16.12%를 설명할 수 있었다. 두 주성분을 조합하면 52.92%까지 설명이 가능하였다.
국내 가시상추 집단은 비교적 높은 유전적 다양성을 가지고 있는 것으로 분석되었다(Fig. 2). 광양 수집종과 부산 수집종은 같은 그룹을 형성하였으며, 대구, 안동 및 청주 수집종이 같은 그룹을 형성하였다. 당진과 김제 수집종은 두 그룹의 중간에 위치하였다. 같은 그룹을 형성한 지역은 각각 남부해안가 및 중부내륙의 지리학적 특성을 공유하고 있다. 비슷한 기후 환경이 유전적으로 비슷한 집단을 형성하는 것에 영향을 준 것으로 판단할 수 있다(D’Andrea et al., 2009).
가시상추의 원산지로 알려진 유럽 집단은 조지아 수집종을 제외한 5개 국가의 수집종이 한 그룹을 이루고 있었으며, 중국 수집종은 유럽 수집종과 같은 그룹에 속하는 것으로 분석되었다. 국내 가시상추 수집종은 해외 수집종과 확연히 구분된 그룹을 이루는 것으로 분석되었다. 국내 수집종이 유럽 및 중국의 가시상추 집단과 완벽하게 다른 그룹으로 형성된 것으로 보아(Fig. 2), 국내 가시상추는 유럽 내륙에서 직접 들어오거나 중국을 통한 유입은 아닌 것으로 판단된다. 현실성 있는 유입경로를 추적하기 위해서는 일본, 미국, 극동러시아 등 대한민국을 기준으로 동쪽에 있는 국가의 가시상추에 대한 다형성 분석이 필요하다고 판단된다.
비록 국내 분포 가시상추의 원산지나 유입경로를 정확하게 찾아내지는 못했지만, DNA 기반 추적방법이 생태계교란식물의 원산지 추적에 충분히 적용될 수 있다는 것을 확인하였다. 우리나라에 분포하고 있는 생태계교란식물의 정확한 유전체 정보와 대상 생물의 원산지 혹은 유입경로로 예측되는 지역의 충분한 유전자원 혹은 DNA 염기서열 정보를 확보한다면, 보다 더 정확하게 추적할 수 있을 것으로 예상된다. 이러한 원산지 정보는 대상 생태계교란식물의 생물학적 방제원 탐색 등에 도움을 줄 것으로 판단된다.