서론
한련초(Eclipta prostrata)는 아시아 원산의 국화과(Compositae) 일년생 식물이다 (Esther Oluwatoyin and Odunayo New Year, 2021). 일반적으로 우리나라를 포함한 열대, 아열대 및 온대 지역에 흔하게 발생한다 (Kim et al., 2017). 우리나라 논 발생 잡초 중 14위 (Ha et al., 2014), 밭 발생 잡초 중 7위를 차지하며 전국에서 발생하고 있다 (Lee et al., 2015). 최근에는 전남지역 옥수수와 콩 재배지에서 우점 상위3위를 차지하며 (Park et al., 2022) 발생이 증가하고 있다. 한편, 한련초는 농경지에 피해를 주는 여러 병원성 진균 및 바이러스 등의 기주로 작용하고 (Murtaza et al., 2018; Qiu et al., 2022), 최근 중국에서는 Acetolactate 합성 저해 제초제에 저항성이 보고되어 (Li et al., 2017) 농작물에 피해를 준다. 한련초와 유사한 종으로 가는잎한련초(Eclipta alba)가 있지만 가는잎한련초가 농작물에 미치는 피해에 관한 연구는 미흡하다.
한련초는 아시아 원산이며 (Chung et al., 2017), 가는잎한련초는 북아메리카, 중앙아메리카 및 남아메리카 원산이다 (Chung et al., 2017; Kaneyama et al., 2024). 과거에는 잡초의 원산과 생태형에 따라 New World (남북 아메리카)에 발생한 직립형의 가는잎한련초와 Old World (유럽, 아시아, 아프리카)에 발생한 포복형의 한련초를 구분하였다 (Saggoo and Kaur, 2010). 하지만 두 종 사이 형태적 특성이 불분명하고, 형태 특성 차이를 환경에 의한 변이로 인식하는 최근의 분류학적 견해로 가는잎한련초를 한련초의 이명(Synonym)으로 분류하고 있다 (Koyama and Boufford, 1981). 이에 따라 중국에서는 한련초와 가는잎한련초를 같은 종으로 분류하고 있다 (He et al., 2022). 또한 인도에서는 계절적인 변화에 따라 E. alba를 erect, prostrata 그리고 intermediate 3가지 형태형으로 분류한다 (Saggoo and Kaur, 2010). 이러한 분류학적 견해와 반대하여 일본에서는 한련초 및 가는잎한련초의 종자와 잎의 형태학적 차이를 기반으로 두 종을 구분하기도 하였다 (Umemoto and Koyama, 2007). 우리나라에서는 종자의 날개 유무와 잎의 길이를 이용하여 두 종을 구분하고 있다 (Kim and Park, 2009). 이처럼 한련초 및 가는잎한련초는 넓은 환경에서 생존하여 다양한 표현형 변이를 가지고 있기 때문에 형태적 분류만으로 두 종을 구분하는 것은 쉽지 않다 (Saggoo and Kaur, 2010) . 따라서 외부 형태학적 분류 이 외에도 해부학적 형태를 확인하거나 또는 이화학적 분류와 DNA 마커를 통한 분자생물학적 분류에 관한 연구가 이루어졌다 (Wahyuni et al., 2023; Muruganantham et al., 2009; Kim et al., 2017) . 특히 DNA 마커를 통한 분자생물학적 분류는 종을 빠르고 정확하게 동정할 수 있기 때문에 한련초와 가는잎한련초 같이 형태학적으로 동정하기 어려운 분류군의 종 분류에 활발하게 이용되고 있다 (Lee et al., 2015).
여전히 잡초 분야에서는 한련초와 가는잎한련초를 구분하여 분류하고 있다 (Kim and Park, 2009). 하지만 우리나라의 국립생태원에서도 최근의 분류학적 견해에 근거하여 가는잎한련초를 한련초의 이명으로 처리하고, 침입외래식물에서 제외하였다 (Ryu et al., 2017). 농가의 경제성을 높이기 위한 효율적인 잡초 관리에서 잡초의 정확한 분류는 필수적이다 (Lee et al., 2015). 특히 한련초는 농경지에서 발생이 확대되고 있는 종이기 때문에 두 종에 대한 명확한 종 분류의 기준이 필요한 상황이다. 따라서 본 연구는 국내 발생하고 있는 한련초 및 가는잎한련초의 형태학적인 차이와 분자생물학적인 차이 비교를 통해 명확하게 두 종을 분류하고자 하였다.
재료 및 방법
식물재료
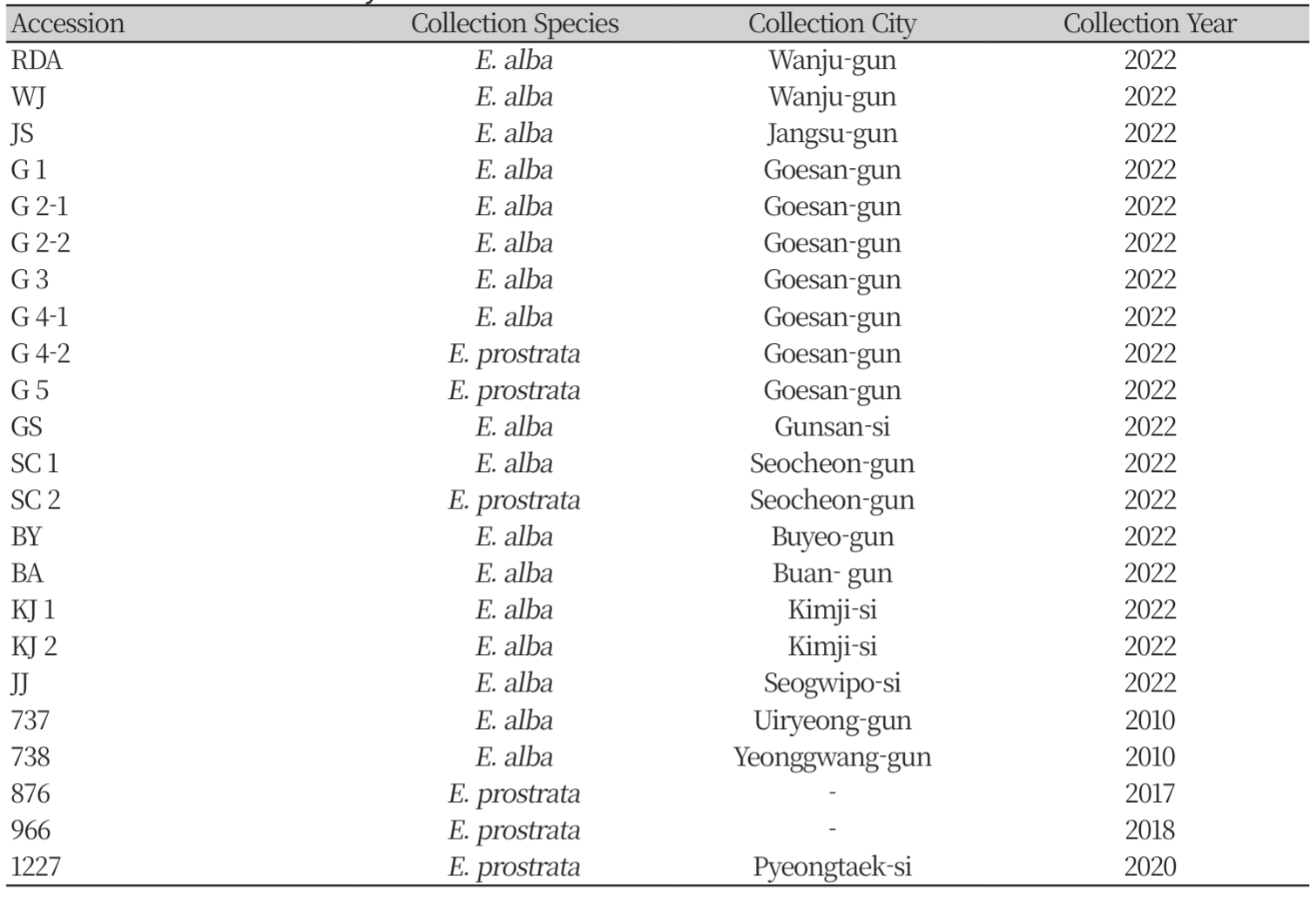
본 연구에서는 총 23 수집종(국립농업과학원에서 보존중인 5 수집종과 2022년 전국에서 수집한 18 수집종)의 한련초 및 가는잎한련초 종자를 사용하였다(Supplementary Table 1).
형태학적 특성 분석
종자의 형태학적 특성 분석: 실체현미경(Stemi 508, Carl Zeiss, Germany)을 이용하여 수집종별 15립의 종자 이미지를 확보했다. iWorks 2.0 (Lanoptik, China)를 사용하여 종자의 가로와 세로 길이를 측정하였다. 추가적으로 이미지상 종자의 날개(종자 양 옆 평평한 형태의 종피, winged parts, Fig 1-c) 유무로 수집종을 그룹화하였다. 종자의 장폭비는 종자의 세로 길이를 가로 길이로 나누어 계산하였다(Fig 1). SAS(v 7.1, Cary, USA)에서 5% 유의수준으로 T-test와 ANOVA 분석을 이용하여 그룹 및 수집종 간의 차이를 확인하였다. 사후 분석은 Duncan 검정을 이용하였다.
잎의 형태학적 특성 분석: 장폭비를 측정한 후, 종자를 일정한 환경(밤 : 낮 = 20/30oC, 10/14 h)이 유지되는 챔버(LH30CCFL-8CT, Nippon Medical & Chemical, JAPAN)에서 최아시켰다. 최아한 개체는 황토(Hwangtomasato, Tohwangto, Korea) : 원예용 상토(Barokeo, Seoul Bio, Chungbuk, Korea) = 1:1 (v/v)의 토양을 채운 플라스틱 사각포트(14*14*11.5 cm)에 이식하여 유리온실 내에서 생육하였다. 생육 초기에 4번째로 발생한 잎(4엽)과 생육 중기에 8번째로 발생한 잎(8엽)의 세로 길이와 가로 길이를 측정하였다. 잎의 장폭비는 잎의 세로 길이를 가로 길이로 나누어 계산하였다. SAS(v 7.1, Cary, USA)를 사용하여 5% 유의수준에서 ANOVA 분석과 Duncan 검정(사후검정)으로 수집종 간의 차이를 확인하였다.
분자생물학적 특성 분석
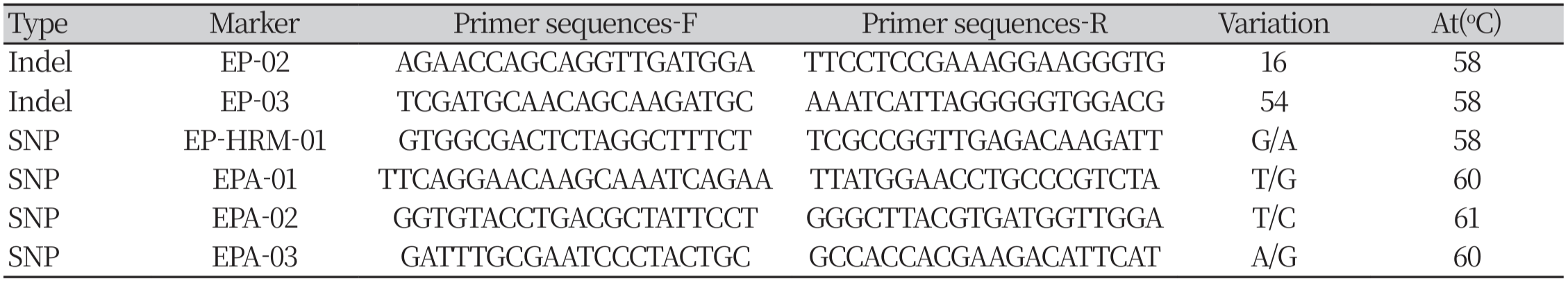
막자사발을 이용하여 온실에서 발아한 한련초 및 가는잎한련초 개체를 액체질소와 함께 갈았다. 모든 개체의 DNA는 DNeasy® Plant Mini Kit (QIAGEN, Germany)를 이용하여 제작자가 제공한 메뉴얼에 따라 추출하였다. 분자생물학적 차이 확인을 위해 기존 연구에서 개발된 Indel 마커 2종와 SNP 마커 1종을 사용하였다 (Kim et al., 2017). 추가적으로 한련초 및 가는잎한련초 엽록체 내 3개의SNP에 대한 마커를 제작하였다(Table 1). DNA 바코드 부위를 증폭을 위해 4 pmol 의 forward primer와 reverse primer 각 2㎕, Inclone® Excel TB 2X Premix 12.5㎕, D.W. 10㎕, 그리고 DNA 1㎕의 혼합물을 제작하였다. T-100 thermal cycle(T100 Thermal Cycler, BIO-RAD, USA)를 사용하였으며, DNA 증폭을 위한 조건은 Supplementary Table 2에 제시하였다.
모든 PCR 산물은 Sanger sequencing으로 양방향 분석되었다. 분석된 염기서열은Mega-11 (v11.0.13, Pennsylvania State University, USA)로 바코드 부위의 multiple sequence alignment을 수행한 후 중첩된 부위에서 오류를 제거하여 최종 염기서열을 결정하였다.
개체들 간의 유연관계 파악을 위해서 Indel 마커(EP_02, EP_03)와 SNP 마커(EP_HRM_01, EPA_01, EPA_02, EPA_03)에 대한 염기서열을 동일한 길이로 자른 후, 순서대로 연결하였다. 연결된 염기서열로 Mega-11를 사용하여 neighbor-joining 방식의 계통도를 작성하였다 (Tamura et al., 2021). 추가적으로 분자생물학적 데이터를 이용하여 종자와 잎의 장폭비를 그룹화한 뒤, 그룹 간에 유의한 차이가 있는지 확인하였다. 통계처리는 SAS(v 7.1, Cary, USA)를 사용하여 5% 유의수준에서 T-test를 하였다.
결과 및 고찰
형태학적 차이
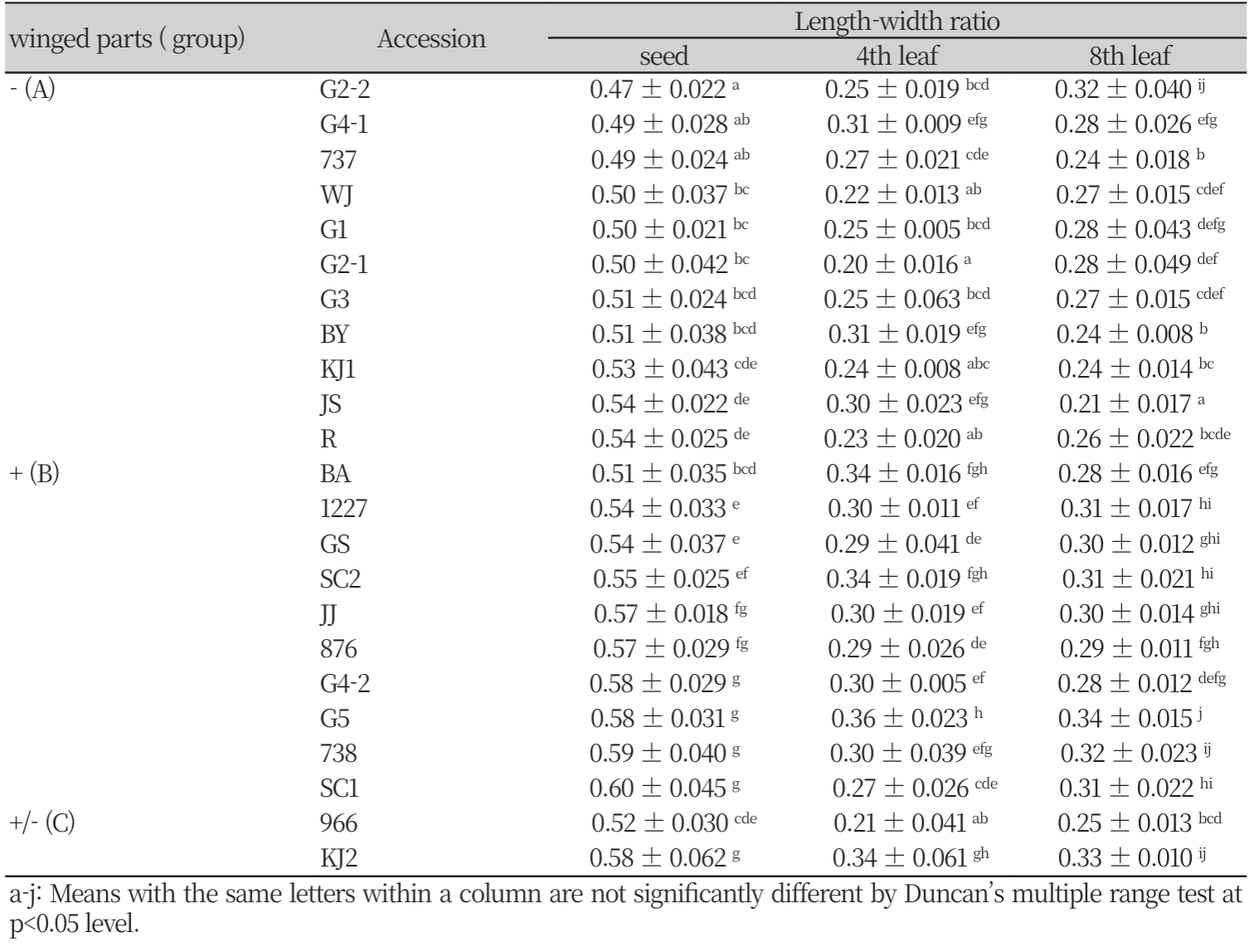
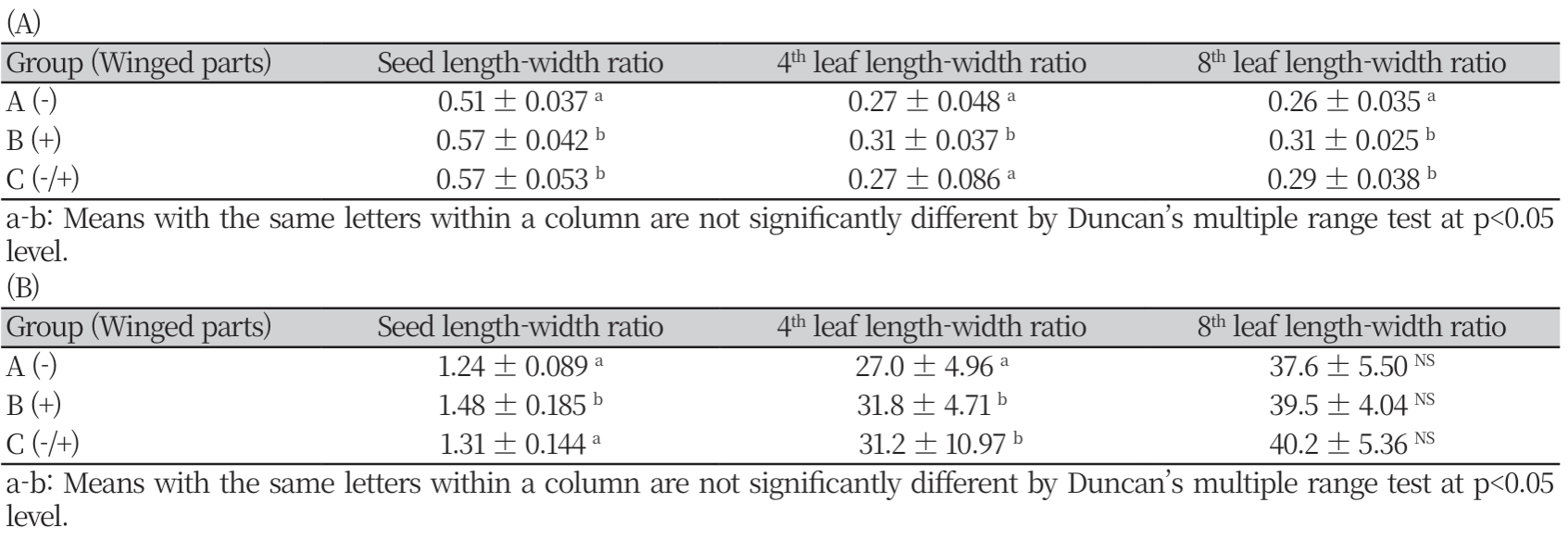
종자의 형태학적 차이: 전체 개체의 종자 장폭비 평균은 0.54±0.034였으며, 수집종의 평균은 0.47±0.022 (G2-2)에서 0.60±0.045 (SC1)까지었다. ANOVA 검정결과 유의하게 차이가 있었지만, Duncan 검정 결과 완전하게 두 그룹으로 나눠지지 않고 10개의 그룹을 형성하며 연속성을 보였다(Table 2). 종자의 날개 유무 기준으로 전체 수집종을 종자 날개가 없는 A, 종자 날개가 있는 B, 두 형태가 모두 포함되어 있는 C 그룹으로 나누었다. 세 그룹의 종자 장폭비 평균은 각각0.51 ±0.037, 0.57±0.042 그리고 0.57±0.053 이었다. B 그룹의 장폭비 평균은 A 그룹보다 높은 값을 보였다. ANOVA 검정 결과 A그룹과 B 그룹은 유의한 차이가 있었다. C그룹은 A그룹과 유의한 차이를 보였지만, B 그룹과는 유의한 차이를 보이지 않았다(Table 3-A). 종자의 장폭비가 아닌 종자의 가로 길이의 평균은 A그룹 1.24±0.089 cm, B그룹 1.48±0.185 cm 그리고 C그룹 1.31±0.144 cm였다. ANOVA 검정 결과, A그룹과 B 그룹은 유의한 차이가 있었다. C그룹의 경우 장폭비 결과와 반대로 B 그룹과 유의한 차이를 보였지만, A 그룹과는 유의한 차이를 보이지 않았다(Table 3-B).
잎의 형태학적 차이: 전체 수집종의 4엽 장폭비 평균은0.28±0.032 였으며, 수집종별 4엽 장폭비 평균은 0.20±0.016 (G2-1)에서0.36±0.023 (G5)까지었다(Table 2). 8엽의 장폭비 평균은0.28±0.021였으며, 수집종별 8엽 장폭비 평균은 0.21 ±0.017 (JS)에서 0.34±0.015 (G5)까지었다(Table 2). 4엽과 8엽의 장폭비 역시 ANOVA 검정결과 유의하게 차이가 있었지만, Duncan 검정 결과 완전하게 두 그룹으로 나눠지지 않았으며 각각 15그룹, 14개의 그룹을 형성하며 연속성을 보였다. 종자의 날개 유무로 분류한 세 그룹의 잎의 장폭비는 4엽에서 0.26±0.048 (A), 0.31±0.037 (B), 0.27±0.086 (C) 였으며, 8엽에서는 0.26±0.035 (A), 0.31±0.025 (B), 0.29±0.038 (C) 였다. ANOVA 검정 결과, 4엽과 8엽 모두에서 A그룹과 B 그룹은 유의한 차이를 보였다. C 그룹의 경우 4엽에서는 B 그룹과, 8엽에서는 A 그룹과만 유의한 차이를 보였다(Table 3-A). 잎의 가로 길이의 평균은 4엽에서 27.0±4.96 cm (A), 31.8±4.71 cm (B), 31.2±10.97 cm (C) 였다. 8엽의 잎 가로 길이 평균은 37.6±5.50 cm (A), 39.5±4.04 cm (B), 40.2±5.36 cm (C) 였다. ANOVA 검정 결과, 4엽의 가로 길이는 A그룹과 B 그룹이 유의한 차이가 있었다. C 그룹은 A그룹과 유의한 차이를 보였지만, B 그룹과는 차이를 보이지 않았다. 반면에 8엽의 가로 길이는 그룹 간의 유의한 차이가 없었다(Table 3-B).
분자생물학적 차이
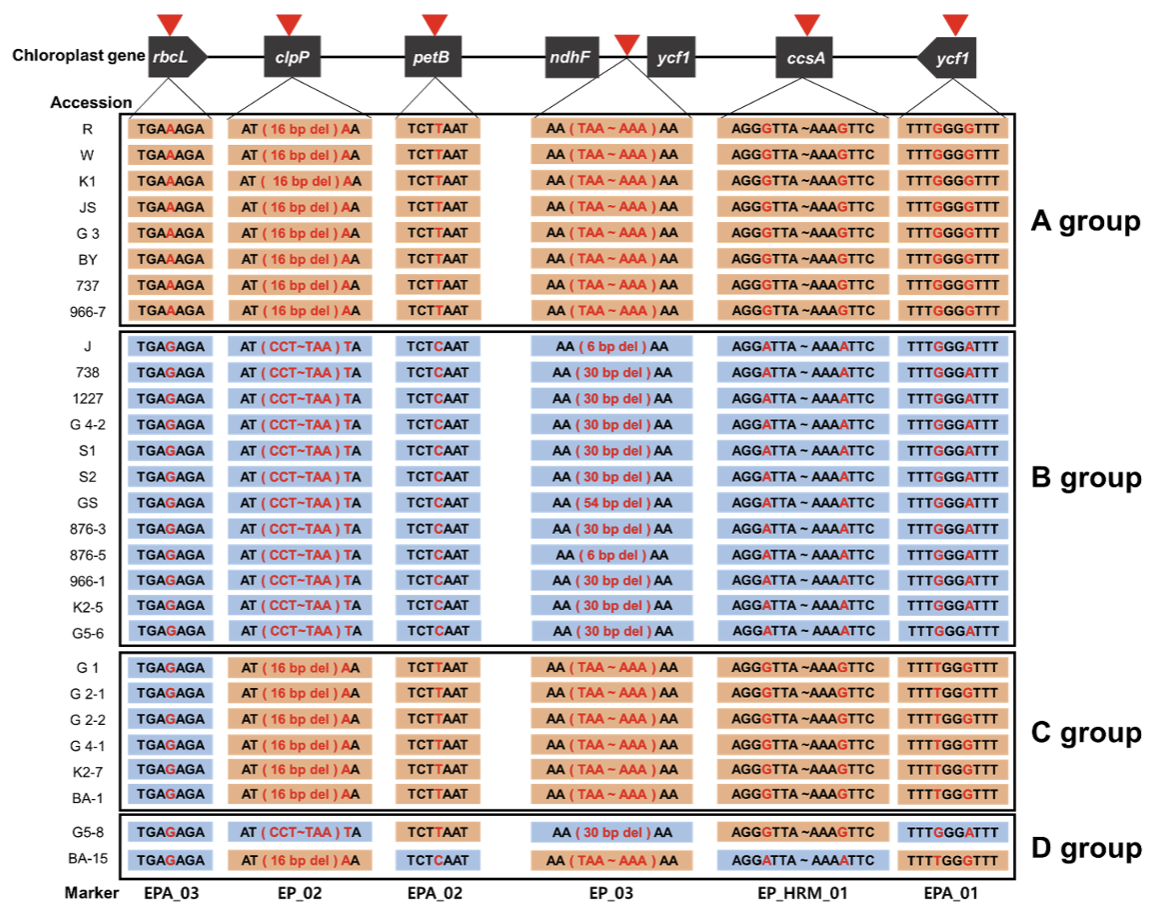
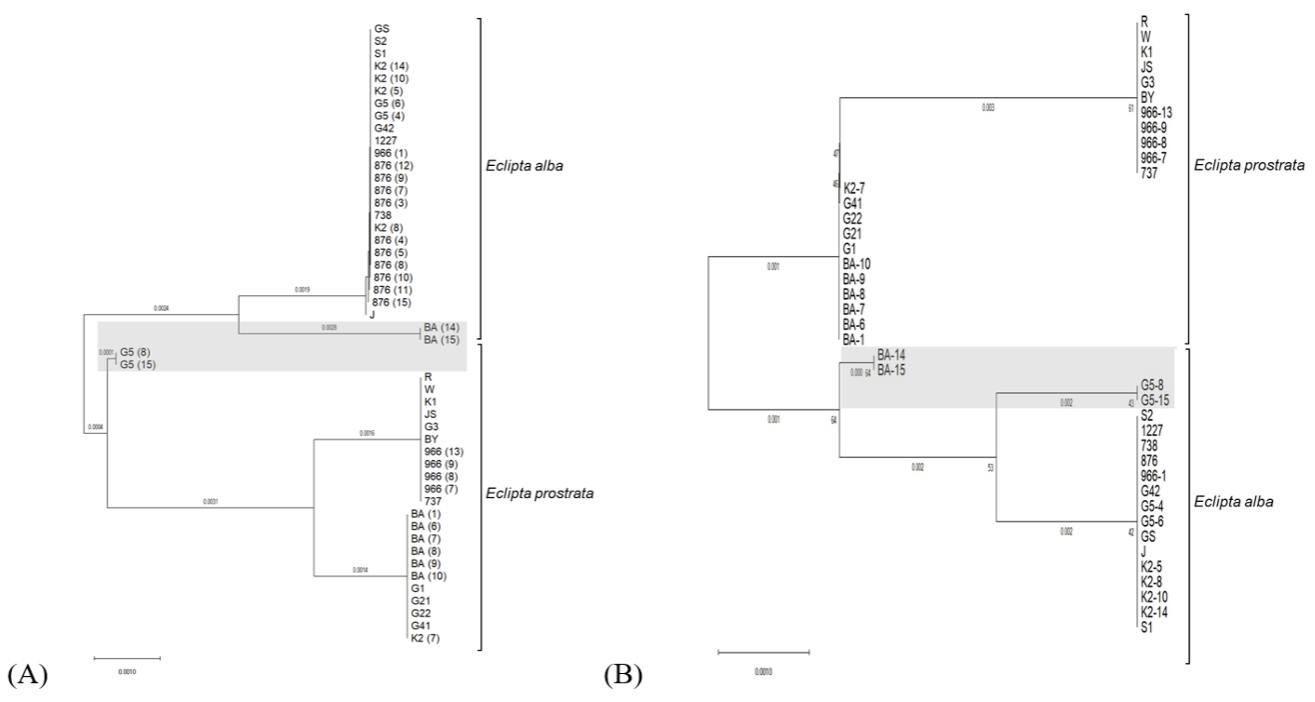
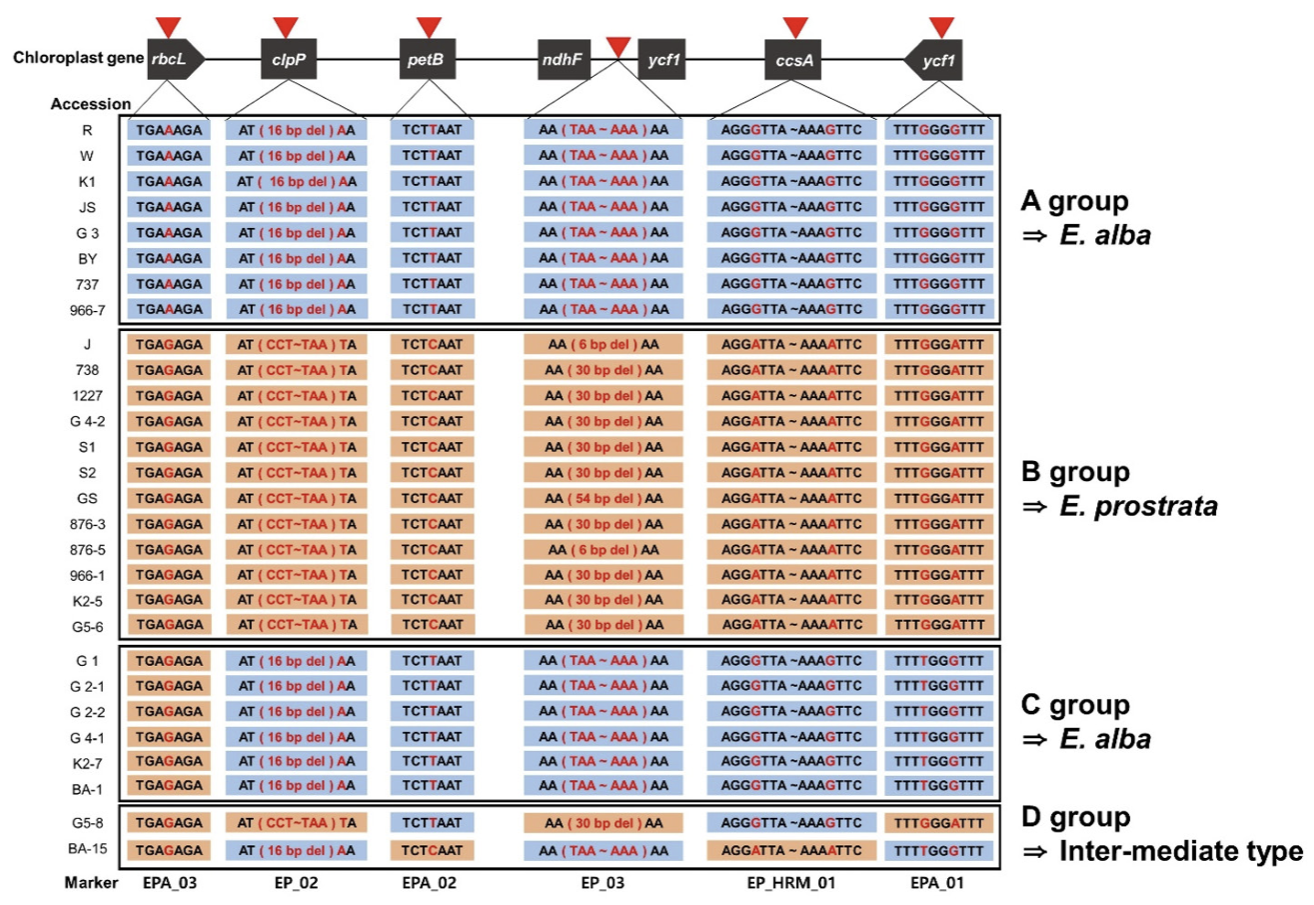
전체 수집종은 6개의 DNA 마커에서 모두 Indel과 SNP 차이를 보였다. 이는 Kim et al. (2017) 연구 결과에 따라 한련초와 가는잎한련초로 분류된다(Fig 2). 전체 수집종을 분류 양상에 따라 네 개의 그룹으로 구분하였다. 전체 23 수집종 중 15수집종, A그룹과 B그룹은 DNA 마커 6종을 통해 각각 가는잎한련초와 한련초로 분류되었다. C 그룹의 경우 5종의 DNA 마커에서 한련초로 분류되었지만, 1종의 마커인 EPA_03(SNP, rbcL region)에서 가는잎한련초로 분류되었다. D 그룹은 같은 수집종(G5, BA) 내 일부 개체에서 2종의 마커(EP_HRM_01, EPA_02)에 대해 염기서열의 차이를 보였다. 그 결과, 한 개체를 마커에 따라 서로 다른 종으로 분류하였다. 결과적으로 본 연구에서 사용된 마커 중 2종의 Indel 마커(EP_02, EP_03)와 1 종의 SNP 마커(EPA_01)만 모든 개체에 대해 같은 종 분류 결과를 나타냈다. 추가적으로 수집종 2종(966, K2)은 같은 수집종 내 한련초와 가는잎한련초가 모두 존재하였다.
6종의 마커 중 1종의 Indel 마커(EP_03)는 deletion 염기의 개수(6, 30, 54 bp) 차이를, 1종의 SNP 마커(EPA_01)는 SNP 개수(1, 2 bp) 차이를 보였다. 6 종의 DNA 마커에 부위를 이용한 계통도 분석 결과, deletion과 SNP의 수준 차이가 아닌 발생 유무에 따라 한련초와 가는잎한련초로 나누어졌다(Fig 3-A). 또한 D 그룹은 다른 수집종과 유전적으로 차이를 보였다.
수집종 내 염기서열의 차이를 보이는 마커 3종(EP_02, EPA_02, EPA_03)을 이용한 계통도 분석에서는 D 그룹은 모두 한련초와 유전적으로 가까웠다(Fig 3-B). 한편 SNP 마커 3종에서 2 종은 아미노산상의 차이가 없었지만, 1종(EPA_02)에서는 E. alba는 류신(leucine), E. prostrata는 세린(serine)으로 아미노산 상의 차이가 있음을 확인하였다.
고찰
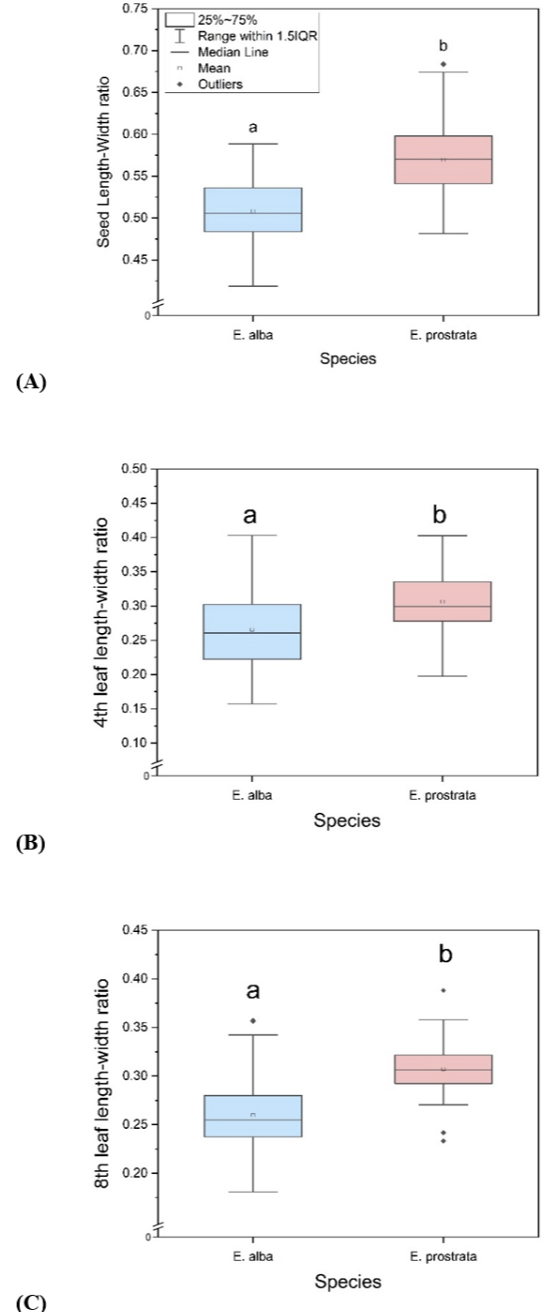
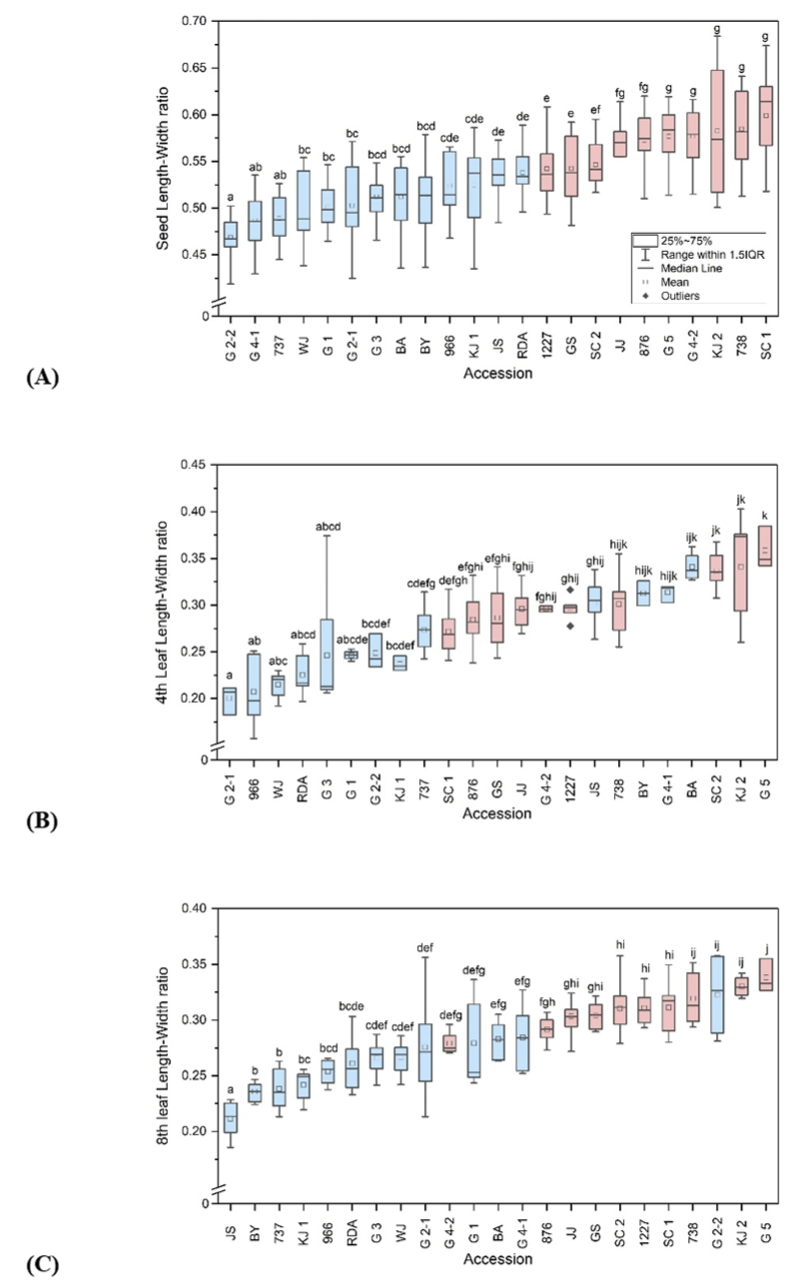
본 실험에서는 국내에서 발생하는 한련초 및 가는잎한련초의 명확한 분류를 위해 형태학적, 분자생물학적 차이를 확인하고자 하였다. 이전의 연구에서 사용된 엽록체 마커 6종 중 한련초의 국내 분포종을 구분할 수 있는 DNA 마커는 3종(EP_02, EP_03, EP_HRM_01)이었다. 그 외 3종 중 1종은 판별도가 낮았으며, 2종은 안정성을 제고하기 위해 타켓 시퀀스에 대해 본 연구에서 재제작하였다(EPA_01, EPA_02). 그 결과 형태학적 특성과 분자생물학적 특성을 모두 이용한 분류는 잎의 형태학적인 특성만을 이용한 Kim et al. (2017) 연구의 결과와 상반되게 한련초와 가는잎한련초를 분류하였다(Fig 4). 본 실험에서는 잎과 종자의 형태학적 특성과 염기서열 상의 특성을 모두 사용하여 한련초와 가는잎한련초를 구분하였다(Fig 5). 그 결과, 종자 장폭비의 경우 7번째 그룹을 기점으로 한련초와 가는잎한련초로 나누어짐을 확인할 수 있었다. 반면, 잎의 장폭비의 경우 4엽과 8엽 모두에서 종자의 장폭비에 비해 완벽하게 두 그룹으로 나누어지지 않았다. 또한 장폭비 데이터를 분자생물학적 분류 결과로 그룹화한 후 t-test를 한 결과, 종자와 잎 모두에서 유의하게 구분되었다(Fig 6). 추가적으로 종자와 잎의 가로 절대값을 이용해 그룹화한 결과, 종자에서는 유의하게 두 그룹으로 구분되었지만, 잎의 경우 4엽과 8엽 모두에서 유의성이 인정되지 않았다(Supplementary table 3). 또한 종자 날개 유무를 기준으로 나누었을 때, 두 형태를 모두 가진 그룹(C그룹, 966과 KJ2)은 분자생물학적으로도 두 종이 모두 확인되었다. 이러한 결과는 형태학적 특성으로 두 종의 추정이 가능하지만, 이전의 연구와 유사하게 잎으로만 두 종을 분류하는 것은 종 구분에 혼동될 수 있으며 종자의 형태학적 차이를 이용할 때 두 종을 더욱 정확하게 분류할 수 있음을 시사한다. 식물에서 종자의 모양 등 형태 특성은 분류학적 진단에 유용하며, 이는 식물의 외부형질 중 종자가 환경에 의한 변이가 적은 것이 원인일 것으로 추정된다 (Song et al., 2015; Kim et al., 2015). 수집종에 대해 형태학적인 특성만을 이용하여 분류한 결과는(Supplementary Table 1) 형태학적, 분자생물학적 특성 모두를 이용한 보다 명확한 분류 결과와 73.91% 일치하였다. 이처럼 형태학적 특성과 분자생물학적 특성을 모두 사용하였을 때 단독 특성을 사용할 때보다 종 분류의 신뢰도가 높아짐을 확인했다. 이러한 결과는 정확한 종의 분류를 위해서는 분자생물학적인 분류와 형태학적인 분류를 모두 사용해야 함을 시사한다.
Fig. 5
Comparison of seed (A), 4th leaf (B), 8th leaf (C) length-width ration of total accessions. Using molecular biological data, blue group is Eclipta alba, red group is E. prostrata. Using morphological data, the same letters within figures are not significantly different by Duncan’s multiple range test at p<0.05 level.
수집종 내 염기서열 차이를 보인 D 그룹에 대해 계통도 분석을 한 결과, 이 그룹은 종간 교배로 인한 중간종(intermediate-type) 일 것으로 추정된다(Fig 4). 이와 유사하게 이전의 Kim et al. (2017)의 연구에서는 두 종의 유전자형을 모두 포함하고 있는 샘플에 대해 확인하였다. 대부분의 국화과 식물은 타가수정을 한다 (Kambli et al., 2022). 국화과 잡초인 한련초 역시 곤충을 매개로 타가수정을 한다 (Perić and Rilak, 2017). 또한 가는잎한련초의 염색체 수는 2n=22 이다 (Bala and Gupta, 2013). 한련초는 대부분의 연구에서 2n=22로 확인된다 (Salamah et al., 2018). 이처럼 한련초와 가는잎한련초의 염색체 수는 동일하며 종간 교배가 가능하다. 두 종은 생활 환경을 공유하며, 유전적 거리가 매우 가깝기 때문에 종간 교배가 일어날 확률이 높을 것으로 추정된다 (Kim et al., 2017). 한편, 한련초와 유사한 E. thermalis와 가는잎한련초의 자연 교배로 추정되는 중간 개체들이 일본에서 보고되었고, 두 종의 교잡종은 종자의 형태에서 두 종의 특성을 모두 보이는 중간적인 형질을 가지고 있었다 (Kameyama et al., 2024). 본 실험의 t-test 분석 결과에서도, 중간종으로 추정되는 개체는 다른 수집종들과 차이를 보이지 않았다 (data was not showing). 따라서, 중간종에 대한 결과를 포함한 두 종의 정확한 분류를 위해서는 실험에 사용된 6종의 마커를 사용해야 함을 시사한다.
식물의 엽록체 유전체는 안정적으로 존재하기 때문에 rbcL을 포함한 여러 종류의 분자 마커가 개발되어 종 판별에 사용되고 있다 (Kim et al., 2022). 본 실험의 분자생물학적 분석 결과, 대부분의 수집종은 두 종으로 구분되었다. 종자의 날개 유무로 분류한 결과와 비교하였을 때, EPA_03 (rbcL) 마커를 이용한 분류 결과와 81.56% 일치하며 6종의 마커 중 가장 낮은 일치성을 보였다. EP_02 (clpP), EP_03 (ycf1-ndhF), EPA_01 (ycf1) 마커는 97.77% 일치하며 6종의 마커 중 높은 일치성을 보였다. 최근 ycf1는 다양한 DNA 바코드 마커 중 rbcL에 비해 국화과 식물을 포함한 대부분의 육상식물에서 높은 다양성을 가지며 차세대 범용 DNA 바코드 마커로 제안되고 있다 (Dong et al., 2015). 본 실험에서도 EPA_01 (ycf1)는 2개의 SNP를 나타내며 3가지의 다형성(한련초: GGGA, 가는잎한련초: GGGG/GTGGG)을 보였다. 따라서 한련초와 가는잎한련초의 명확한 분류에 사용되는 6종의 DNA 마커 중 높은 효율성을 위해 1종의 마커를 선발한다면, EPA_01 (ycf1)의 사용이 가장 합리적이라고 생각된다.
한편, 본 실험에 사용된 모든 마커는 반성유전인 엽록체 기반이기 때문에 잡종이 모계로 동정되어 분류가 왜곡될 수 있다 (Kim et al., 2022). 따라서 양친의 유래를 확인할 수 있는 리보솜 내 ITS 영역 등 (Jin et al., 2018) 핵 기반의 마커를 통해 이를 보완하는 연구가 필요할 것으로 생각된다.
명확한 종의 구분은 잡초 관리 및 방제 측면에서 중요한 기초 정보이다. 따라서 본 연구와 같이 형태학적, 분자생물학적 차이를 이용하여 종을 구분하는 방법은 한련초와 가는잎한련초 같이 형태 특성이 유사한 잡초 종의 명확한 분류에도 유용하게 사용될 것으로 판단된다.