서 언
가뭄, 저온, 고온, 고염분(high salt), 홍수와 같은 환경스트레스는 식물의 생장과 적응에 장애요인으로 작용하고, 식용, 사료용, 공업용, 조경용 등을 포함한 모든 작물의 품질 저하 및 농업 생산성 감소의 주요 원인이 된다(Mahajan and Tuteja, 2005; Saini and Westgate, 2000; Smirnoff, 1998). 따라서 작물육종에서 환경스트레스에 대한 내성을 높이는 일은 중요한 육종 목표가 된다. 특히 잔디처럼 경관이 중요한 조경식물은 사람들이 밟으며 이용하는 공원, 정원, 스포츠 공간, 골프장 등에서 푸르름을 유지해야 하므로 잔디관리 비용을 최소화하기 위해서는 식용작물 이상으로 환경스트레스에 대한 강한 내성이 요구된다.
일반적으로, 스트레스 내성 품종은 전통육종 방법에 의해서도 육성될 수 있지만 교잡이 가능한 유전자원의 한계가 있으며, 보다 우수한 품종을 육성하기 위해서는 스트레스에 대한 식물의 반응 메커니즘을 분자적인 수준에서 이해함으로써 유용유전의 발굴 및 형질전환이 가능한 생명공학기술이 필요하다(Mahajan and Tuteja, 2005; Zuo et al., 2019; Zuo et al., 2020; Zuo et al., 2021). 식물들이 생장과 적응에 저해가 될 수 있는 스트레스에 노출되면 해당 스트레스를 인지함과 동시에 분자적 신호에 변화가 생긴다. 이러한 분자신호에 따라 환경스트레스에 반응하는 많은 유전자들이 전사 수준에서 조절되고, 이 유전자들의 산물이 최종적으로 스트레스에 대한 식물의 저항성을 높여 주고 건강한 상태가 유지되도록 한다(Shinozaki and Yamaguchi-Shinozaki, 2000; Shinozaki et al., 2003). 식물이 환경스트레스에 저항하기 위해서는 신호전달유전자, 효소유전자, 전사조절유전자 등을 포함한 다양한 종류의 유전자가 관여하는 것으로 알려져 있다(Chen et al., 2002; Cushman and Bohnert, 2000). 환경스트레스에 반응하는 대표적인 전사조절인자로는 DREB/CBFs, AREB/ABFs, NACs (NAM, ATAF, and CUC) 등이 알려져 있다(Hu et al., 2006; Ito et al., 2006; Jeong et al., 2010; Kim et al., 2004; Kizis and Pages, 2002; Xue et al., 2011). 그 중에서 NAC family 유전자의 단백질은 N-말단 쪽에 NAC 도메인을 가진 식물 특이적인 전사인자로서, 애기장대에서 약 117개, 벼에서는 약 151개의 유전자가 존재하고 있는 super family 전사인자 유전자이다(Fang et al., 2008; Nuruzzaman et al., 2010). 애기장대에서 가뭄에 반응하는 RESPONSIVE TO DEHYDRATION 26 (RD26) 유전자가 보고된 이후로 페튜니아에서 배아발생 동안 줄기생장점의 형성과 꽃의 분열조직에서 기관 시원체의 위치결정에 중요한 역할을 하는 NO APICAL MERISTEM (NAM) 유전자가 보고되었다(Souer et al., 1996). 또한 ATAF subfamily에 속하는 NAC 유전자들은 여러 종류의 식물에서 저온, 고염, 가뭄 등의 스트레스 또는 잎의 노화와 관련 있는 것으로 보고되었다(Ohnishi et al., 2005). 애기장대의 CUC1과 CUC2는 꽃과 배아의 발달에서 기관의 분리를 조절하는 NAC 유전자로 보고되었고, 이 때부터 이 유전자 단백질들에서 공통적으로 N-말단에 보존된 영역을 NAC (NAM, ATAF1, CUC2) 도메인이라고 하였고 이 도메인을 가진 유전자를 NAC 유전자라고 명명하였다(Aida et al., 1997; Takada et al., 2001; Vroemen et al., 2003). 밀에서 노화를 조절하는 TtNAM-B1 유전자는 종자단백질과 아연과 철의 함량을 증가시킨다(Uauy et al., 2006). 벼의 STRESS-RESPONSIVE NAC1(SNAC1)은 가뭄 스트레스 하에서 전사수준이 증가하며, SNAC1을 과발현시킨 벼 형질전환 식물체는 대조구에 비해 가뭄과 염스트레스에 대한 저항성이 현저하게 높다고 보고되었다(Hu et al., 2006). 밀에서 분리된 TaNAC69는 가뭄 스트레스 조건에서 전사가 상향조절되었고 이 유전자를 밀에 과발현 시켰을 때 가뭄 스트레스에 대한 저항성이 증가하는 것으로 보고되었다(Xue et al., 2011). 이처럼 식물의 NAC family 유전자는 식물의 발달뿐만 아니라 여러 가지 환경스트레스에 대한 저항성과 관련되어 있다.
크리핑 벤트그래스(Agrostis stolonifera L. cv. Penncross)를 비롯한 한지형 잔디들은 고온 스트레스에서 잎과 뿌리 등의 생장장애를 일으키며 스트레스가 길어지면 잎의 노화가 진행되며 결국 고사하게 된다(Duff and Beard, 1975; Huang et al., 1998). 이러한 고온 스트레스는 온도 자체의 피해뿐만 아니라 급격한 물의 증발로 인한 물 부족을 수반하며 이로 인해 이차적으로는 병원균에 대한 저항성을 약화시킬 수 있다(Tester and Bacic, 2005). 본 연구는 작물에 각종 피해를 일으키는 환경 스트레스에 대한 저항성을 증가시켜 줄 수 있는 연구와 함께 유전자 자원을 확보하기 위한 일환으로 크리핑 벤트그래스에서 환경스트레스에 반응하는 NAC family 유전들을 분리하기 위해 수행되었다.
재료 및 방법
식물재료
본 연구는 한지형 잔디인 크리핑 벤트그래스(Agrostis stolonifera L. cv. Penncross)를 사용하였으며 21℃, 장일조건(16 h light/8 h dark)의 식물생장실에서 관리되었다.
RNA 추출 및 1차 cDNA의 합성
RNA를 분리하기 위한 잎 재료는 시료채취 후 바로 액체질소에 급속 냉각시켰다. 시료를 미리 냉각시킨 막자사발에 넣고 액체 질소로 완전한 분말형태가 될 때까지 분쇄시켰다. RNase-free 1.5 mL micro tube에 TRI reagent (MRC, Cincinnati, OH, USA) 1 mL을 미리 넣고 분쇄된 시료를 넣어 혼합한 후 total RNA를 추출하였다. 추출된 RNA의 품질은 1% 아가로스 겔에 1 μg을 전기영동을 통해 확인하였다. RT-PCR을 위한 1st strand cDNA는 1 μg의 RNA를 0.5 μg의 oligo dT primer, 5x M-MLV reaction buffer, 10 mM dNTP, 25 unit RNasin Ribonuclease inhibitor (Promega, Madison, USA), 200 unit M-MLV reverse transcriptase (Promega), DEPC dH2O를 혼합한 후 42℃에서 1시간 반응으로 합성하였다.
cDNA 유전자 단편의 클로닝
ClustalX 프로그램(http://www.clustal.org/clustal2/)을 이용하여 애기장대 NAC 단백질들의 아미노산 서열을 비교한 다음 상호 간의 상동성이 높은 N-말단 가까운 부위와 C-말단 가까운 부위에서 아미노산 서열을 기반으로 4개의 degenerate DNA primer를 제작하였고 PCR을 통해 NAC family cDNA들의 무작위 증폭을 유도하였다(Table 1). 증폭된 DNA는 pGEMT easy T 벡터(Promega)에 클로닝하였다.
AsNAC 단편 유전자들의 상동성 분석
AsNAC 단편 유전자들의 상동성을 분석하기 위해 기존에 보고된 몇몇 NAC 유전자 정보를 NCBI Genbank 데이터베이스를 통하여 수집하였다. 수집된 유전자 서열들과 본 연구에서 분리한 AsNAC cDNA 단편 유전자들을 FASTA format으로 정렬한 다음 ClustalX 프로그램을 통하여 분석한 후 MEGA 프로그램(https://www.megasoftware.net/)을 통하여 Neighbor joining방법으로 Phylogenetic tree를 작성하였다.
mRNA 발현 분석을 위한 스트레스 처리
mRNA 발현분석을 위해 이용된 크리핑 벤트그래스는 지름 15 cm, 높이 20 cm의 화분에서 온도, 습도, 광조건을 각각 21℃, 60%, 12 h로 일정하게 조절되는 생장실에서 재배하였고 이 식물체를 이용하여 스트레스를 처리하였다. 고온 (heat)스트레스는 38℃, 60%, 12 h light에서 하루에 한 번씩 38℃의 물을 관수시키면서 처리하였다. 암(dark) 스트레스는 3 mM의 MES (2-(N-morpholino)ethanesulfonic acid) 완충용액에 1년생의 크리핑 벤트그래스잎을 담근 후 암 스트레스 조건(21℃, 60%, 24 h dark)에서 노화를 진행시켰다. 기내(in vitro)에서 자란 크리핑 벤트그래스에서 노화된 잎과 노화되지 않은 잎의 발현양의 차이를 보기 위해 크리핑 벤트그래스의 종자의 종피를 제거한 후 100% EtOH와 2% NaOCl로 살균한 후 MS배지에 파종하였다. 파종된 크리핑 벤트그래스는 25℃에서 장일조건(16 h light/8 h dark)으로 성장시켰으며 18주 후에 노화가 진행된 잎과 진행되지 않은 잎을 각각 분리하여 샘플을 채취하였다. 각각의 스트레스에 노출된 잎을 채취하여 -70℃에 보관 후에 total RNA를 추출하였다. 추출된 total RNA는 스트레스에 따른 발현을 보기 위해 1 μg의 RNA를 cDNA로 합성하였다.
결과 및 고찰
크리핑 벤트그래스에서 환경스트레스에 유도되는 NAC 유전자의 분리
크리핑 벤트그래스에서 환경스트레스에 의해 유도되는 AsNAC 유전자들을 분리하기 위해 애기장대 NAC family 유전자들의 단백질 상동성을 비교하여 아미노산 서열의 보존영역(conserved domain)을 확인하였다. 그리고 그 보전영역의 아미노산에서 유추된 적절한 degenerate primer들을 합성하여 PCR을 통해 무작위로 후보 유전자군의 증폭을 유도하고자 하였다. Degenerate primer와 PCR을 이용한 family 유전자들을 클로닝하는 기술은 Preston (2003)에 의해 소개되었다. 본 연구는 4-cutter 제한효소로 DNA를 절단하는 아이디어를 고안하여 무작위로 혼합된 PCR 산물에서 독립된 유전자들을 선별하였다. 애기장대에서 NAC 단백질들은 모두 5개의 서브도메인(subdomain)으로 구성된 NAC 도메인을 가지고 있다(Fig. 1A). Degenerate primer는 Fig. 1A에 제시된 그림에서 A 서브도메인에서 정방향(forward direction)으로 NAC F1과 NAC F2를 제작하였고 D 서브도메인에서 역방향(reverse direction)으로 NAC R1과 NAC R2를 제작하였다(Table 1). Fig. 1B는 degenerate primer로 NAC family 유전자들을 분리하기 위한 아이디어를 요약한 그림이다. 고온 처리된 잎과 노화된 잎에서 추출한 RNA에서 합성된 1차 cDNA를 주형으로 4가지 조합으로 PCR을 진행하여 각각 예측된 크기(약 0.4 kb)의 증폭된 DNA를 얻었다(Fig. 1C). 증폭된 DNA는 PCR 산물을 plasmid T-vector에 클로닝하여 35개의 클론을 얻었다(Fig. 1C). 이들 중 중복된 클론들을 제거하기 위해 4개의 염기(GTAC)를 인식하는 RsaI 제한효소로 후보 클론 DNA들을 절단한 다음 아가로스 전기영동 겔 상에서 밴드 패턴을 비교하였으며 염기서열 분석을 통해 최종적으로 총 9개의 독립적인 cDNA 단편 유전자를 확보하였고 각각 AsNAC16, AsNAC17, AsNAC18, AsNAC19, AsNAC20, AsNAC21, AsNAC22, AsNAC23, AsNAC24로 명명하였다. 무작위로 증폭된 DNA의 클론들의 중복 여부는 DNA 염기서열 분석으로도 확인될 수 있지만 비용을 절감하기 위해 1차적으로 제한효소를 이용하여 중복된 클론들을 제거하였다. 4개의 염기서열을 인식하는 제한효소는 통계적으로 256 bp 마다 1개의 인식부위가 기대되므로 400 bp 내에는 1-2개의 절단 가능성 부위가 있을 가능성이 높다. 4개의 염기서열을 인식하는 여러 종류의 4-cutter 제한효소가 있지만 애기장대 NAC 유전자들의 염기서열을 참조하여 RsaI를 선택하였다. Fig. 1C의 우측 사진은 RsaI으로 처리한 DNA 전기영동의 한 예를 제시한 것이다.
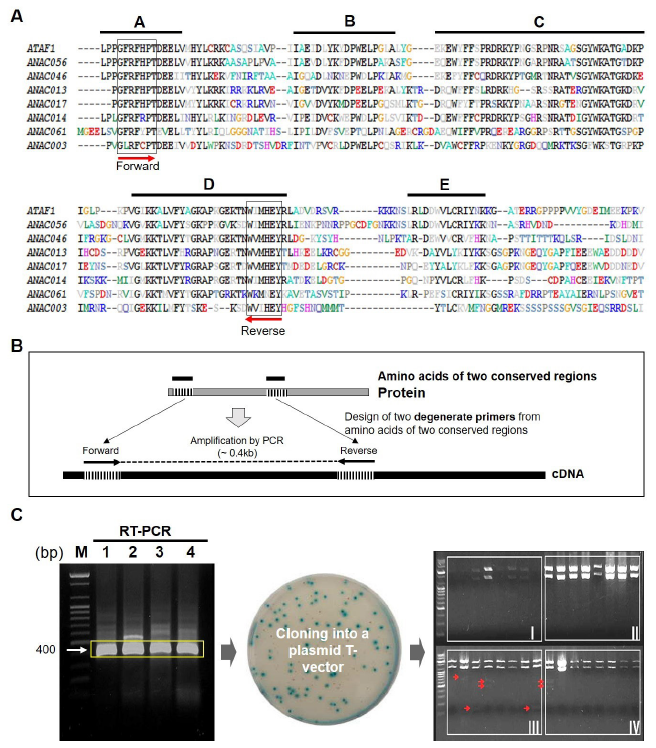
Fig. 1. Isolation of NAC cDNAs from creeping bentgrass using conserved domains of NAC proteins in Arabidopsis thaliana . (A) Comparison of amino acid sequences of NAC domains consisting of 5 subdomains (A to E) from A. thaliana respectively. (B) Experimental scheme to isolate NAC cDNAs of creeping bentgrass by PCR using degenerate primers based on conserved amino acid sequence domains. (C) Cloning and screening of PCR products into a T-plamid vector. Lane 1 to 4 are RT-PCR products. Lane 1 NAC F1 and NAC R1. Lane 2 NAC F1 and NAC R2. Lane 3 NAC F2 and NAC R1. Lane 4 NAC F2 and NAC R2.
AsNAC 유전자들의 단백질 상동성 분석
크리핑 벤트그래스에서 분리한 9개의 AsNAC cDNA의 염기서열을 단백질로 번역한 다음 아미노산 서열을 비교한 결과, 전체적으로 서로 49-98%의 상동성을 나타냈다(Table 2), NAC 서브도메인들의 주요 아미노산들도 잘 보존되어 있는 것으로 확인되었다 (Fig. 2A). 계통발생학적 분석을 통해 9개 AsNAC 단백질들은 특정 아미노산의 차이로 인해 3개의 서브그룹으로 나누어지는 것으로 나타났다(Fig. 2A). 이러한 결과는 기존에 보고된 그룹 I의 Ia-Ic, 그룹 II의 IIa-IIc와 같은 보존된 모티프들에 의해 분류되는 것을 확인하였다(Jeong et al., 2010). 클로닝한 크리핑 벤트그래스의 9개 NAC 단백질들과 기존에 기능이 알려진 다른 식물들의 NAC 단백질들을 가지고 계통발생학적 분석을 수행하였다. 그 결과 AsNAC 단백질은 기능적인 관점에서 크게 두 그룹, 즉 식물발달과 스트레스 반응 단백질로 분류되었다(Fig. 2B). AsNAC16, AsNAC17, AsNAC21, AsNAC22는 ATAF (Lu et al., 2013), SNAC1 (Hu et al., 2006), OsNAC3 (Zhang et al., 2021), OsNAC4 (Kaneda et al., 2009), OsNAC5와 OsNAC6 (Takasaki et al., 2010), TaNAC69-1 (Xue et al., 2011)과 함께 스트레스 조절유전자로 묶였고 AsNAC18, AsNAC19, AsNAC20, AsNAC23, AsNAC24는 NAM (Souer et al., 1996), CUC1과 CUC2 (Takada et al., 2001; Vroemen et al., 2003), OsNAC1, OsNAC2, OsNAC7 (Kikuchi et al., 2000), ONAC300 (Kusano et al., 2005)과 함께 발달 조절유전자로 묶였다(Fig. 2B). 따라서 AsNAC16, AsNAC17, AsNAC21, AsNAC22는 환경스트레스 조절 유전자일 가능성이 높고 식물체에 과발현시켰을 때 스트레스 내성을 향상시킬 수 있는 후보 유전자가 될 수 있을 것으로 기대된다.
AsNAC 유전자들의 발현 분석
크리핑 벤트그래스에서 분리한 AsNAC 유전자들이 고온, 암상태 등의 환경스트레스와 노화 등에 반응하는지 알아보기 위해 각각의 유전자에 대한 특이적 PCR 프라이머를 제작하여 RT-PCR을 수행하였다(Table 1). 상동성 분석에서 스트레스 조절유전자에 속한 유전자 중에 AsNAC16은 노화된 잎에서 발현이 증가하였으나 고온과 암조건에서는 반응하지 않는 것으로 나타났다(Fig. 3B). AsNAC17과 AsNAC22는 고온, 암조건 및 노화된 잎에서 모두 발현이 증가되었다(Fig. 3B). AsNAC21은 고온에서는 발현이 높아졌으나 암조건이나 노화된 잎에서는 큰 차이를 보이지 않았다 (Fig. 3B). 식물발달조절 그룹에 속한 유전자 중에도 AsNAC20과 AsNAC23 외에 AsNAC24, AsNAC18, AsNAC19는 스트레스 또는 노화된 잎에서 발현의 변화가 관찰되었다(Fig. 3B). AsNAC24는 암 처리 7일 후 발현이 증가되었고 AsNAC18은 암 처리 7일 후 발현이 증가되는 반면 노화된 잎에서는 발현이 현저히 감소되었고 AsNAC19는 노화된 잎에서 오히려 발현이 증가되었다(Fig. 3B). 본 연구에서 분리된 크리핑 벤트그래스의 NAC 유전자들은 예상한 대로 환경스트레스에 대한 발현의 변화를 보여주었고, 향후 이 유전자들은 애기장대의 ATAF1 (Liu et al., 2016), 벼의 SNAC1 (Liu et al., 2014), 밀의 TaNAC69 (Xue et al., 2011) 등의 경우처럼 과발현 등의 생명공학적인 방법으로 작물의 스트레스 내성을 향상시키기 위한 후속 연구를 이어 갈 가치가 충분한 유전자 자원이 될 수 있을 것으로 사료된다.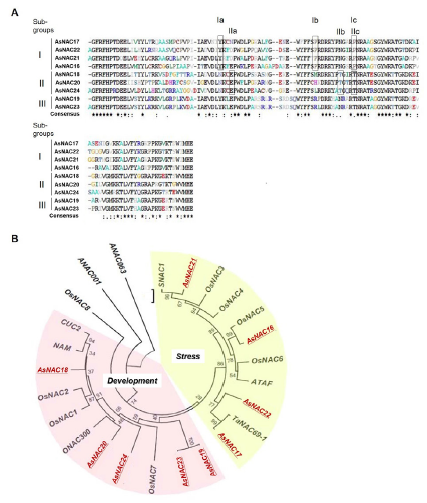
Fig. 2. Alignment and phylogenetic analysis of deduced proteins of partial AsNAC genes isolated from creeping bentgrass. (A) Multiple sequence alignment of AsNACs. Squared boxes show conserved domains of NAC proteins. (B) Phylogenetic relationship of AsNACs with NAC proteins from other plants including Arabidopsis (ANAC001, ANAC063, ATAF, and CUC2), rice (OsNAC1, OsNAC2, OsNAC3, OsNAC4, OsNAC5, OsNAC6, OsNAC7, ONAC8, SNAC1, and ONAC300), petunia (NAM), and wheat (TaNAC69-1).
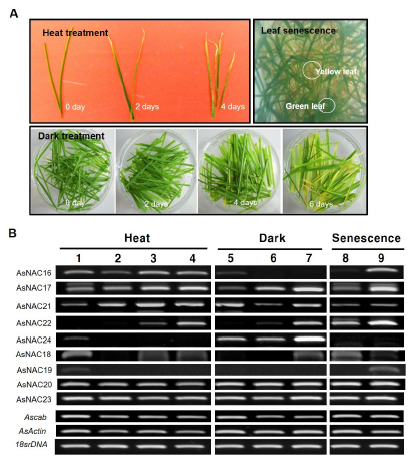
Fig. 3. Expression of AsNAC genes under abiotic stresses. (A) Heat or dark-treated leaf and senescence leaf of creeping bentgrass. (B) Expression of the cloned AsNAC cDNAs by RT-PCR. Lane 1, 2, 3, and 4 represent 0, 2, 4, and 6 days after heat treatment, respectively. Lane 5, 6, and 7 represent 0, 2, and 6 days after dark treatment, respectively. Lane 8 and 9 green and yellow leaves of creeping bentgrass, respectively. Ascab, AsActin, and 18srDNA were used as controls for the experiment.
Authors Information
Bo-Hwa Kim, Agricultural Technology Institute, Jeju Special Self-Governing Province, Researcher
Hyeon-Jin Sun, Subtropical Horticulture Research Institute, Jeju National University, Research professor
Hyo-Yeon Lee, Subtropical Horticulture Research Institute, Jeju National University, Professor
Hong-Gyu Kang, https://orcid.org/orcid.org/0000-0002-2428-2757




