서 언
나비나물속(Genus Vicia)은 콩과(Fabaceae)의 콩아과(Faboideae)에 속하며, 전 세계적으로 180-210종이 분포하고 있는 것으로 추정하고 있으며(Hanelt and Mettin, 1989), 우리나라에는 22종이 국가표준식물목록(www.nature.go.kr/kpni)에 등록되어 있다. 우리나라 농경지 100대 잡초에 속하는 나비나물속 잡초로는 살갈퀴(Vicia angustifolia), 새완두(V. hirsuta), 얼치기완두(V. tetrasperma)로 3종이 포함된다(Lee et al., 2017).
국내 농경지에서 발생하는 나비나물속 잡초는 전국적으로 고르게 분포하는 것으로 알려져 있다(Cho et al., 2009). 살갈퀴의 발생량이 전국적으로 가장 많았으며, 새완두와 얼치기완두는 중북부 지역보다 남부지역의 발생량이 더 많았다. 헤어리베치(V. villosa)와 얼치기완두 등과 같은 나비나물속 식물들은 잡초 발생 억제, 토양 물리성 개선 및 양분공급 등으로 인해 일부 하계작물 농경지에서 피복작물이나 녹비작물로 이용되기도 한다(Cho et al., 2009). 나비나물속 식물들은 도로변이나 과수원 등으로 종자가 퍼져 잡초화하는 등의 문제를 발생시키기도 한다. 효율적이고 효과적인 잡초관리를 위해서는 정확한 종 동정이 필수조건이다. 이론적으로 나비나물속의 잡초는 잎, 꽃, 종자 등의 크기나 모양으로 구분이 가능하지만, 자연교잡 등의 원인으로 중간형질을 보이는 경우가 많아(Hollings and Stace, 1974), 잡초방제의 ‘골든타임’이라고 할 수 있는 유묘기에 동정이 매우 어렵다.
Hebert et al. (2003)에 의해 제안된 DNA 바코드는 특정 유전자의 단편 염기서열을 비교 분석하여 종 수준에서 빠르고 정확하게 동정할 수 있는 방법이다. 선충의 경우에는 미토콘드리아 DNA의 COII (Cytochrome OxidaseII)에서 16sRNA 사이의 염기서열을 이용해 종을 동정하기도 하며(Ko et al., 2017), 진균 에서는 ITS (internal transcribed spacer)부위의 염기서열을 사용한다(Xu, 2016). 그러나 잡초를 포함한 고등식물에서는 엽록체 DNA의 rbcL과 matK 염기서열을 조합하여 이용하는 것을 권고하고 있으며(Hollingsworth et al., 2009), 분류군에 따라 핵산 DNA의 ITS 부위나 엽록체 DNA의 psbA-trnH 염기서열 등을 활용하고 있다(Chen et al., 2010). 이러한 DNA 바코드를 이용하여 형태적으로 구분이 어려운 종을 구분하는 시도가 있으며(Kim et al., 2018; Lee et al., 2015), 국가적 생물다양성 보전 측면에서 활용하는 경우도 있다(Kress, 2017).
따라서 본 연구는 농경지에서 발생하는 나비나물속 잡초를 대상으로 바코드 유전자로 알려진 엽록체 DNA의 rbcL의 단편 염기서열과 핵 DNA의 ITS 부위의 염기서열을 분석하여 국내 나비나물속 잡초의 유전적 다양성을 확인하고, 분자생물학적 분류 및 동정에 활용 가능성을 판단하고자 하였다.
재료 및 방법
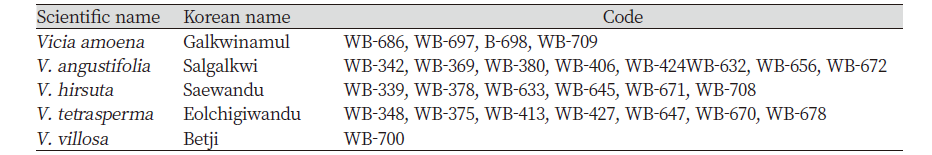
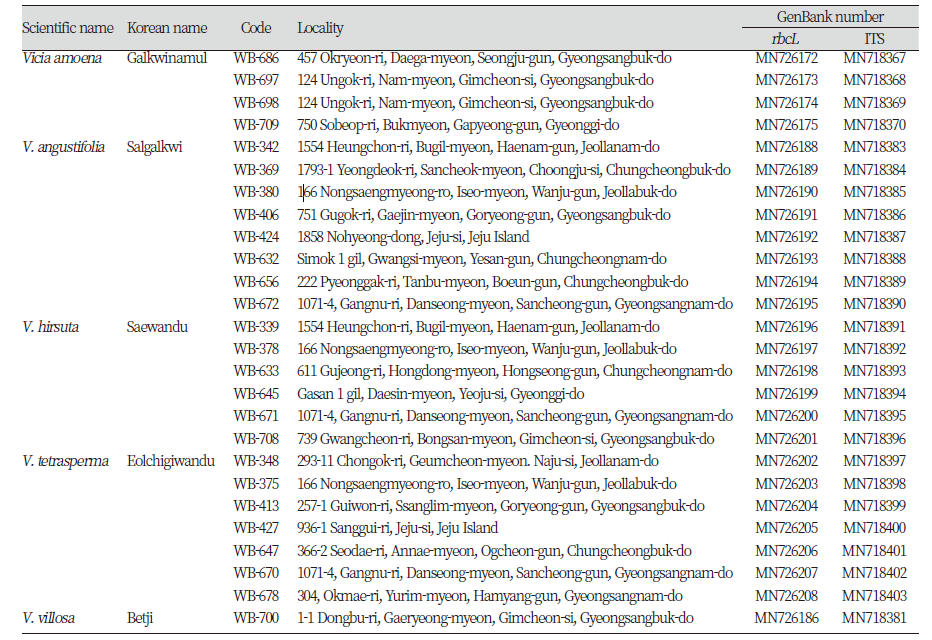
경기도 2지역, 충청도 5지역, 전라도 7지역, 경상도 11지역, 제주도 1지역 등 논둑 및 밭둑을 포함한 전국의 농경지에서 나비나물속(Vicia spp.) 5분류군 26생태형을 채집하여 본 연구에 사용하였다(Table 1 and Supple. Table 1). 채집한 시료의 잎을 분리한 후, 액체질소와 막자사발을 이용하여 곱게 분쇄하였다. 분쇄된 시료에서 DNeasy Plant Mini Kit (QIAGEN, Hilden, Germany)를 이용하여 제공되는 프로토콜에 따라 DNA를 추출하였다. 엽록체 DNA의 rbcL 유전자 및 ITS 부위의 부분 염기서열을 비교하기 위해 Table 2의 프라이머를 사용하였다. PCR은 Heo et al. (2019)의 방법을 참고하였다. genomic DNA 0.5 μL, 10 mM의 dNTP 0.4 μL, 25 mM의 MgCl2 1 μL, 5 x Green GoTaq® Flexi Buffer (Promega Co., Madison, USA) 4 μL, 4 pmole의 forward/reverse primer 각 1 μL 그리고 0.5 unit의 GoTaq® Flexi DNA polymerase (Promega Co., Madison, USA.) 0.1 μL를 혼합하여 95℃에서 2분, 35회의 95℃에서 20초(denaturation), 55℃ (rbcL) 혹은 53℃ (ITS)에서 40초(annealing), 그리고 72℃에서 45초(extension)의 조건으로 PCR하였다. PCR 산물은 QIAGEN QIAquick® PCR Purification Kit (QIAGEN, Hilden, Germany)를 이용하여 정제하였다.
바코드 부위의 염기서열은 BioEdit 7.2.6과 MEGA 6.06을 이용하여 분석하였다. 분석된 바코드 구간에서 나비나물속 잡초의 종내 및 종간변이율은 Kimura 2-Parameter distance model을 이용하여 산출하였다. 미국 국립생물공학정보센터(NCBI)에 등록된 나비나물속의 rbcL 염기서열 16개(미국 5개 및 중국 11개) 및 ITS 염기서열 23개(일본, 이란 각각 2개, 팔레스타인 4개 독일, 뉴질랜드, 중국 각각 5개)와 국내 수집종의 rbcL 및 ITS 염기서열을 비교하고 Neighbor-joining (NJ) 계통도(Saitou and Nei, 1987)를 작성하여 상관관계를 분석하였다.
결과 및 고찰
rbcL 및 ITS 구간에서 나비나물속 잡초의 종내 및 종간변이율
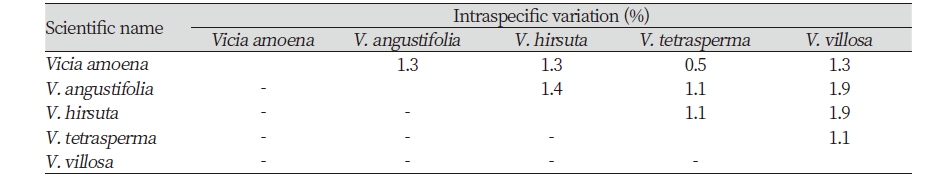
rbcL 및 ITS 구간에서 나비나물속 잡초의 종내변이율(intraspecific variation)은 Table 3과 같다. 채집지역이 지역적으로 분산되어있음에도 불구하고, 나비나물속잡초의 rbcL 구간에서 종내변이율은 갈퀴나물(Vicia amoena)만 0.1%였으며 나머지 4종에서 종내변이는 없었다. ITS 구간에서 종내변이율은 갈퀴나물이 0.6%, 살갈퀴(V. angustifolia)가 0.1%였으며, 나머지 3종에서 종내변이는 없었다. 마디풀과 잡초의 종내변이율은 rbcL구간에서 0.00-0.13%로(Heo et al., 2019) 나비나물속 잡초의 종내변이율과 같이 매우 낮았다. 따라서 rbcL과 ITS, 두 구간의 염기서열은 국내 나비나물속 잡초 4종 각각 고유하다고 할 수 있다.
|
Table 3.Intraspecific variation (%) of rbcL partial sequences and internal transcribed spacer (ITS) region sequences in Vicia spp. 
|
rbcL 구간에서 나비나물속 잡초의 종간변이율(interspecific variation)은 Table 4와 같다. 나비나물속잡초의 rbcL 구간에서 종간변이율은 평균 1.3%였다. 종간변이율은 새완두와 헤이리베치(V. villosa) 그리고 살갈퀴와 헤어리베치에서 1.9%로 가장 컸으며, 갈퀴나물과 얼치기완두에서 0.5%로 가장 작았다. ITS 구간에서 나비나물속 잡초의 종간변이율은 Table 5와 같다. 종간변이율은 평균 3.9%였으며, 살갈퀴와 헤어리베치에서 4.7%로 가장 컸고, 갈퀴나물과 얼치기완두에서 3.0%로 가장 작았다. rbcL과 ITS 구간의 염기서열 중 종간변이율은 ITS 구간에서 더 큰 것으로 나타났으며, 공통적으로 살갈퀴와 헤어리베치의 차이가 가장 컸고, 갈퀴나물과 얼치기완두가 가장 작았다. 본 연구에서 사용된 염기서열은 모두 단편 염기서열이기 때문에, 살갈퀴와 헤어리베치의 유연관계가 가장 멀고 갈퀴나물과 얼치기완두의 유연관계가 가깝다고 할 수 없다. 다만, 두 염기서열 모두 종내변이율이 매우 낮고 종간변이율이 크다는 것을 통해 그 구간이 종을 구별하는 특이적 염기서열임을 알 수 있다. 따라서 rbcL 및 ITS 구간의 염기서열 분석을 통해 나비나물속 잡초를 분류할 수 있으며, 형태적 특성이 발현되기 전인 유묘기 때에도 효율적으로 분류할 수 있음을 시사한다.
|
Table 5. Interspecific variation (%) of internal transcribed spacer (ITS) region sequences in Vicia spp. 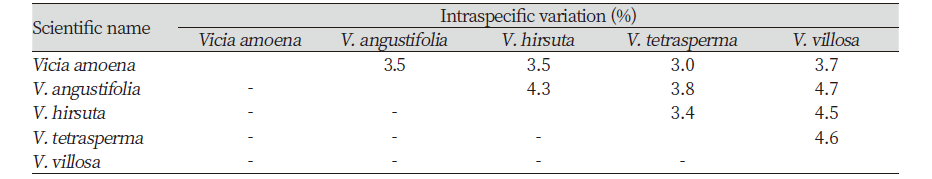
|
종내 유연관계
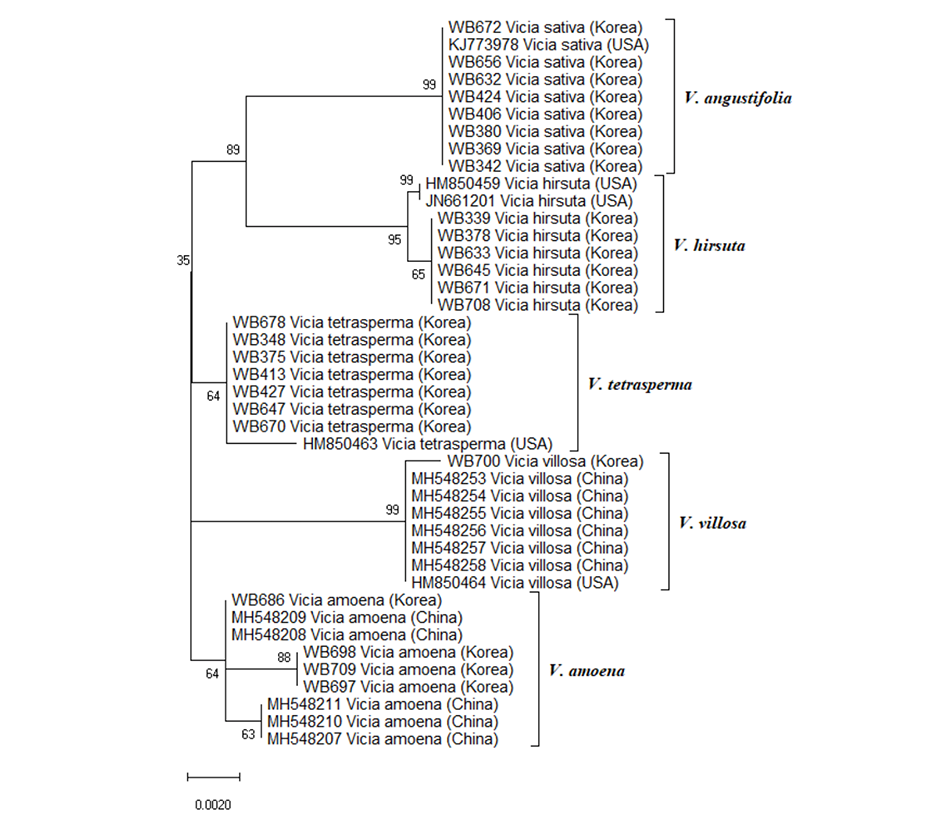
국내 5종의 나비나물속 잡초 중, 갈퀴나물(V. amoena)에서만 rbcL 단편 염기서열에서 변이가 있었다(Table 3). 중국 갈퀴나물 5 수집종의 단편염기서열(Wu et al., 2020)과 같이 분석한 결과 3가지 그룹으로 나뉘었다(Fig. 1). 국내 새완두와 얼치기완두는 각각 미국 수집종과 염기서열의 차이를 보였고, 국내 헤어리베치는 중국과 종내변이가 있는 것으로 나타났다. 살갈퀴(syn. V. sativa)는 미국 수집종과 차이를 보이지 않았다.
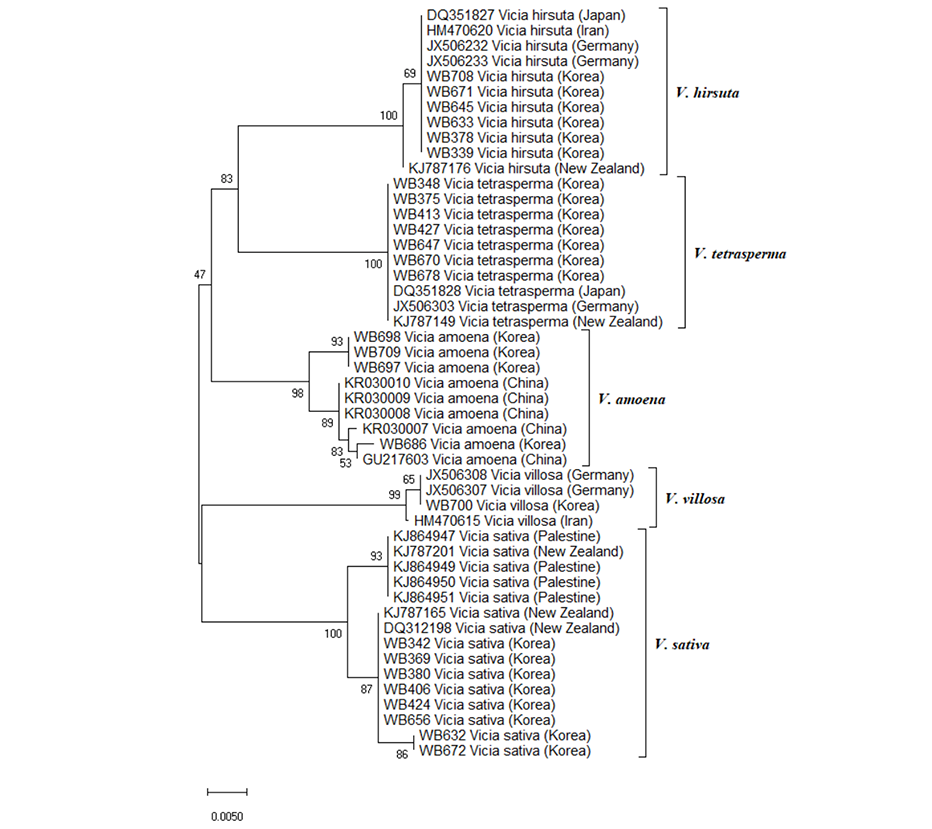
ITS 구간의 염기서열을 이용한 NJ (Neighbor-joining) 분석 결과, rbcL보다 다양한 변이를 보였지만, 나비나물속의 잡초를 명확하게 분류하였다(Fig. 2). 새완두는 일본, 이란, 독일 및 뉴질랜드의 분석결과와 비교한 결과 뉴질랜드 수집종을 제외한 모든 국가의 수집종이 하나의 그룹으로 분류되었다. 얼치기완두는 일본, 독일 및 뉴질랜드와 함께 분석한 결과 하나의 그룹으로 분류되었다. 갈퀴나물은 rbcL의 경우와 마찬가지로 크게 3그룹으로 분류되었다. 국내 수집 헤어리베치는 독일과 한 그룹에 속했으며, 이란 수집종과는 다르게 분류되었다. 팔레스타인 및 뉴질랜드의 수집종과 비교한 살갈퀴의 경우, 한국의 살갈퀴는 크게 두 그룹으로 나뉘었으며, 뉴질랜드 수집종의 일부와 같은 그룹으로 묶였다.
rbcL과 ITS 염기서열을 종합적으로 비교했을 때, 100대 잡초인 새완두, 얼치기완두 및 살갈퀴보다 갈퀴나물의 유전적 다양성이 컸다(Fig. 1 and Fig. 2). 갈퀴나물은 저온에서도 잘 자라는 식물로 알려져 있으며, 지역 및 고도에 따라 유전적 다양성이 풍부한 것으로 알려져 있다(Liu et al., 2013). 갈퀴나물 수집종은 경북 내에서도 의성(WB697, 698)과 성주 수집종(WB686)이 다르게 분류되었다. 충분한 수집종의 염기서열을 확보하여 분석한다면 풍부한 유전적 다양성을 확보할 수 있을 것으로 판단된다.
rbcL 및 ITS의 단편 염기서열을 이용하여 NJ 계통도를 작성한 결과, 나비나물속의 살갈퀴, 새완두, 얼치기완두, 헤어리베치, 갈퀴나물은 명확하게 분류되었다(Fig. 1 and Fig. 2). 최근, Wu et al. (2020)은 161종의 나비나물속 식물의 종 분류에는 matK 유전자 혹은 trnH-psbA 구간의 염기서열이 가장 효율적이고, ITS 구간은 보조적인 역할로 사용할 때 효과적이라고 보고하였다. 또한 Raveendar et al. (2015)은 ITS2 구간과 matK 유전자의 염기서열을 동시에 이용했을 때, 나비나물속 식물의 분류가 효과적이라고 주장했다. 그러나 국내 농경지에서 발생하는 나비나물속 잡초의 종류가 제한적이고, 동계작물 및 과수원에서 제한적으로 발생하는 점을 감안했을 때, rbcL 단편 염기서열의 분석 만으로도 충분한 변별력을 가지고 있으며, 간이성과 활용성은 두 방법과 비교해 더욱 높다고 평가할 수 있다.
본 연구에서 rbcL 단편 염기서열과 ITS 구간의 염기서열의 비교를 통해 나비나물속 잡초의 분류를 성공적으로 수행하였다. 개체가 너무 어리거나 중간적인 형태적 특성으로 인해 현장에서 나비나물속 잡초의 동정이 불가능한 경우, rbcL 및 ITS 구간의 염기서열 분석을 통해 정확한 종 동정을 할 수 있을 것이다. 정확한 종 정보는 작물의 재배 특성 등과 함께 효과적이고 효율적인 방제법을 제시할 수 있는 필수적인 정보로 활용되어야 할 것이다.
Acknowledgement
This research was supported by a project from the Cooperative Research Program for Agricultural Science & Technology Development (Project No. PJ012640) of the RDA.
Authors Information
Gyeong-Jong Heo, National Institute of Agricultural Sciences, Master
In-Seong Seo, National Institute of Agricultural Science, Research assistant
Hyun-A Seo, National Institute of Agricultural Science, Research assistant
Jin-Won Kim, National Institute of Agricultural Science, Researcher, http://orcid.org/0000-0002-3245-3704