돼지풀(Ambrosia artemisiifolia L.)은 북아메리카 원산인 일년생 하계잡초이다. 이 잡초의 꽃가루에는 호흡기 알러지 유발 물질이 있기 때문에, 꽃가루 형성시기(8-9월)에는 공공보건을 위협하기도 하며(Wopfner et al., 2005), 빠른 초기생장과 타감물질의 분비로 주변 식생의 생물종다양성을 떨어뜨린다고 알려져 있다(Cowbrough et al., 2003, Vidotto et al., 2013). 이로 인해, 환경부는 식물로는 처음으로 단풍잎돼지풀(Ambrosia trifida)과 함께 돼지풀을 생태계교란생물(invasive alien species)로 1999년에 지정하였다. 1980년대부터 이미 전국에 분포하고 있었고(Yim and Jeon, 1980), 주로 경기도 및 강원도 북부의 농경지에서 돼지풀의 발생이 많은 것으로 조사되었다(Kim et al., 2017). 이미 확산된 생태계교란식물을 화학적으로 방제하는 것은 사용 시기 및 장소 등 현실적 제약이 크기 때문에, 생물학적 방제법을 사용하는 것이 효율적으로 알려져 있으며, 그 사례도 다수 보고되어 있다(e.g. Acacia spp. in the South Africa, Zachariades et al., 2017). 국내 돼지풀에 대한 생물학적 방제법을 개발하기 위해서는 유전적 다양성 확인이 전제되어야 하지만, 국내 돼지풀의 유전적 다양성에 대하여는 보고된 바가 없다. 따라서 본 연구에서는 국내 수집 돼지풀 집단의 유전적 다양성을 평가하고자 하였으며, 이를 통해 생태계교란생물 제거를 위한 생물학적 방제법의 기초자료를 제공하고자 하였다.
실험에 사용된 돼지풀 집단은 국내 5지역(광주, 대구, 사천, 영암 및 청주)에서 수집하였으며, 각 수집지역에서 1 m 간격으로 6~10 개체를 채집하였다. 국내 돼지풀 집단의 유전적 다양성 평가를 위한 대조군으로 러시아 우수리스크(Ussurisk) 및 핫산(Hassan) 지역의 돼지풀 DNA를 사용하였다. 수집한 돼지풀의 신엽을 액체질소와 함께 막자사발을 사용하여 곱게 간 후, DNeasy® plant mini kit (QIAGEN, Hilden, Germany)를 이용하여 DNA를 추출하여 사용하였다.
돼지풀의 유전적 다양성을 검토하기 위해 사용된 SSR 마커는 총 15개였다. 기존 논문 등 문헌검색을 통해 97개의 EST-SSR 마커를 1차 선발하였고, 2차에 걸친 PCR 재현성 검정을 통해 최종적으로 7개의 유효한 SSR 마커를 선발하여 실험에 사용하였다(Table 1). 신규 SSR 마커를 개발하기 위해 돼지풀 gDNA를 NGS한 결과, 총 137,168개의 SSR구역이 발견되었다(data not shown). 이 중 다형성과 재현성이 높고, 실험적 접근성이 좋은 염기서열을 선택하기 위해 repeat size가 21 bp이상인 것, 프라이머와 SSR간의 간격이 30 bp인 것, annealing temperature가 58~60℃사이인 것, 프라이머 크기가 20~25 bp인 것을 종합적으로 고려하여 최종적으로 총 8개의 신규마커를 선발하였다(Table 1).
|
Table 1. Information of 15 primers used in this study, EST-SSR markers and developed markers from NGS analysis. 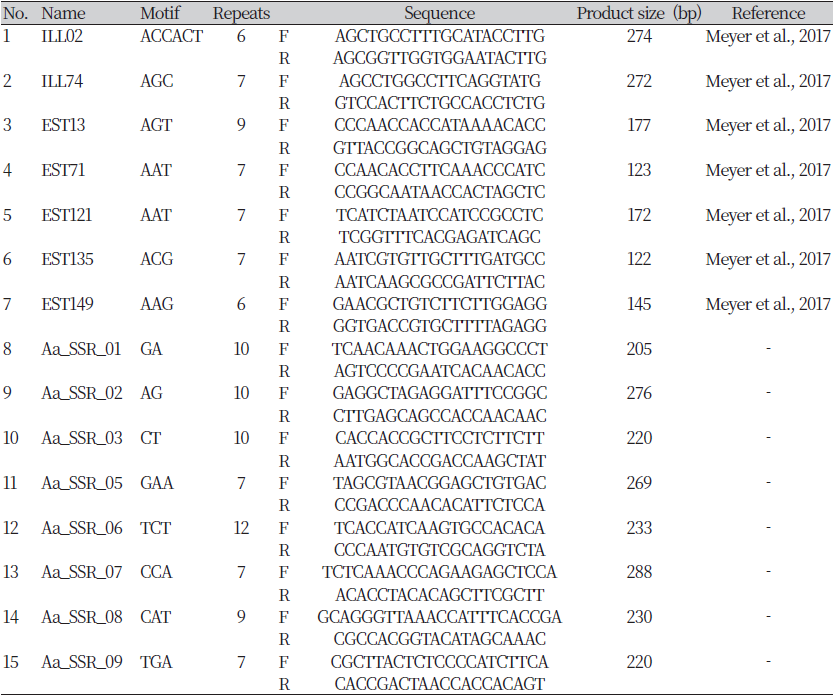
|
15개의 SSR마커를 2개씩 짝지어 총 8개의 그룹을 만들었고, 두 개의 forward primer에 각각 형광표지(6-carboxyfluorescein 및 hexachloro-fluoresceine) 붙여 프라이머를 제작하였다. 돼지풀 gDNA (10 ng μL-1) 1 μL와 PCR cocktail 11.5 μL (형광표지된 프라이머(5 μM each) 1.2 μL, 10X buffer 1.25 μL, dNTPs (2 mM each) 1.25 μL, 및 1.25 u F-Star Taq polymerase (Biofact, Daejeon, Korea))를 혼합하여 12.5 μL의 PCR cocktail을 제조하였다. PCR 조건은 (1) 95℃에서 10분, (2) 95℃에서 30초, 58℃에서 30초, 72℃에서 1분을 30회 반복, (3) 68℃에서 30분이었다. PCR 산물은 ABI3730xl DNA analyzer (Applied Biosystems, Foster, USA)를 사용하여 전기영동한 후, GeneMapper 4.0 (Applied Biosystems, Foster, USA)을 이용하여 PCR product의 길이를 분석하였다. 대립유전자 수(number of different alleles), 이형접합성(heterozygosity), F-statistics 및 주성분분석(principal coordinates analysis; PCoA)은 GenAlEx 6.501을 통해 분석하였다(Peakall and Smouse, 2006).
대조구 포함 전체 돼지풀 7 집단의 유전적 다형성을 ILL02 포함 총 15개의 SSR 마커로 검정한 결과, 대립유전자의 수는 평균 4.8±0.19 였으며, 유효대립유전자 수는 3.3±0.16이었다(Table 2). 관측 이형접합성(HO)은 0.5±0.02였고, 기대 이형접합성(HE)은 0.6±0.02였다. 15개 마커 중 11개에서 HO가 HE보다 작았고, 같은 것은 3개, 더 큰 것은 1개였다(Table 2). 전반적으로 HO가 HE보가 작기 때문에 본 연구에서 평가한 돼지풀 5 집단은 유전적 다양성에 부정적인 영향을 받고 있다고 판단할 수 있었다.
|
Table 2. No. of alleles, no. of different alleles, no. of effective alleles, observed and expected heterozygosity, and fixation index of 15 markers used in this study. 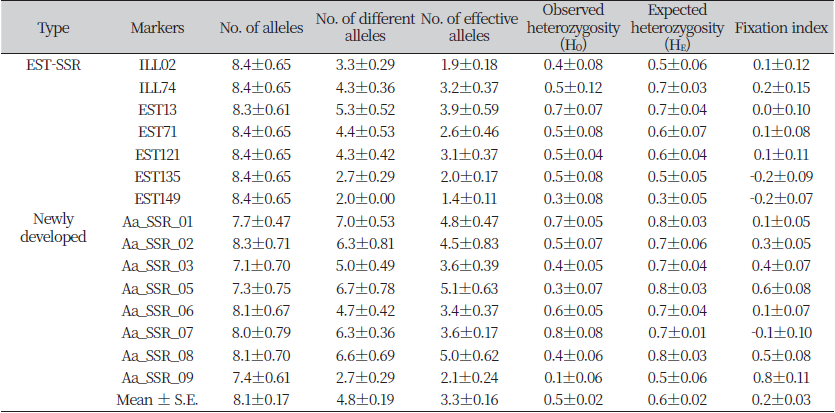
|
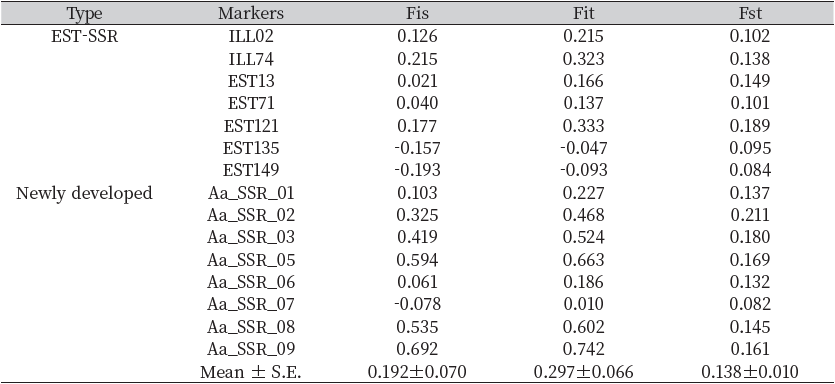
Fst값은 집단 간 유전적 유사도를 나타내는 값으로, 값이 높을수록 집단간 유전적 차이가 크다는 것을 의미한다. F-statistics 분석 결과, 돼지풀 7집단에 대한 15 마커의 Fst 평균값은 0.138±0.010이었다(Table 3). 즉, 연구에 사용된 각 돼지풀 집단들 간에 유전적 차이가 다소 있다고 할 수 있다. 집단 내 유전적 차이를 나타내는 Fis 및 전체 집단에 대한 유전적 차이를 나타내는 Fit값이 각각 0.192±0.070 및 0.297±0.066으로 분석되었다(Table 3). 이는 실험에 사용한 각 돼지풀 집단 혹은 전체에 대한 집단이 유전적으로 차이가 있음을 의미한다. 분석의 결과를 종합해보면, 국내 돼지풀 집단의 다양성이 이 현재에도 다소 높은 수준으로 유지되고 있다고 판단된다.
돼지풀은 일반적으로 바람에 의해 타가수분하는 것으로 알려져 있고, 강력한 자가불화합성(self-incompatibility)이 있기 때문에, 자가수분할 경우 종자생산량이 극도로 줄어든다고 알려져 있다(Friedman and Barrett, 2008). 또한, 제초활동 등으로 인해 일부 돼지풀 집단이 2년만에 소멸하여 개체수준으로 산재하여 나타나기도 하며(Kim et al., 2018), 돼지풀 모니터링 지점의 50%에서 분포면적이 감소한 사실들이 보고되었다(Kim et al., 2022). 본 연구결과와 돼지풀의 수분특성, 그리고 돼지풀 분포면적 감소로 판단했을 때, 현재 국내 돼지풀 집단은 유전적 다양성이 줄어들고 있는 것으로 판단되며, 집단간 혹은 국가간 추가적인 유입은 제한적일 것으로 예상된다. 그러나 집단 내부의 유전적 다양성은 매우 높은 편이기 때문에(Table 3), 건설 등 토양의 인위적 교란 혹은 범람 등 자연적 교란으로 인해 집단의 유전적 다양성이 높아지고, 폭발적으로 발생이 증가할 가능성이 있다. 따라서 특정 집단에 대한 주기적인 모니터링은 지속되어야 할 것이다.
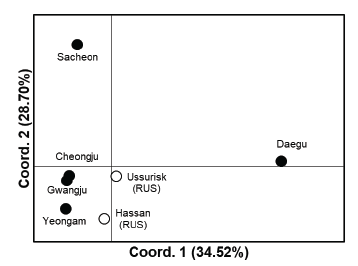
국내 돼지풀 5집단 및 러시아 돼지풀 2집단에 대한 주성분분석한 결과는 Fig. 1와 같다. 제1주성분으로 34.52%를 설명할 수 있었으며, 제2주성분으로 28.70%를 설명할 수 있었다. 두 주성분을 조합하면 63.22%까지 설명이 가능하였다. 청주, 광주 및 영암 집단은 유전적으로 매우 유사한 그룹으로 분석되었으며, 사천과 대구 수집종은 독립적인 그룹으로 분석되었다. 특히 러시아 수집종은 사천 및 대구 수집종을 제외한 국내 수집종과 같은 그룹을 형성하고 있었다. 이를 통해, 국내 일부 돼지풀 집단과 극동러시아 지역의 돼지풀 집단의 유전적 구성이 유사한 것으로 판단되었으며, 상호 연관성이 있다고 추측되었다.